Sistema Unificado para a Criação de um Crânio Composto no OrtogOnBlender XP
Attention
Este material utiliza a seguinte licença Creative Commons: Atribuição 4.0 Internacional (CC-BY 4.0).
Attention
Caso encontre algum erro no texto, sinta-se à vontade para informá-lo aos autores, o contato pode ser feito via redes sociais acadêmicas informadas no início do capítulo.
Introdução
O presente capítulo apresenta a ferramenta unificada de reconstrução de tomografia computadorizada e criação de crânio composto, que está sendo desenvolvida para o OrtogOnBlender XP (OOB_XP), uma versão baseada no Blender 4.2 LTS, inicialmente para o Linux Ubuntu 24.04 LTS e o Windows, com planejamento de expansão para o MacOS.
Como Funciona
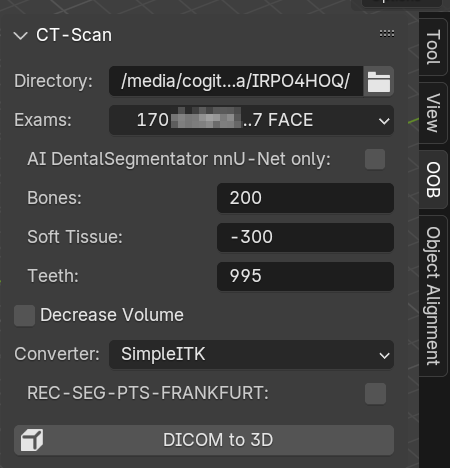
Fig. 17 Etapa inicial da configuração.
O funcionamento da ferramenta é basicamente o mesmo daquele explanado em [B_Moraes_et_al_2024], onde o usuário inicialmente informa o local onde se encontram os arquivos DICOM e posteriormente indica qual exame pretende que seja reconstruído em 3D (Fig. 17).
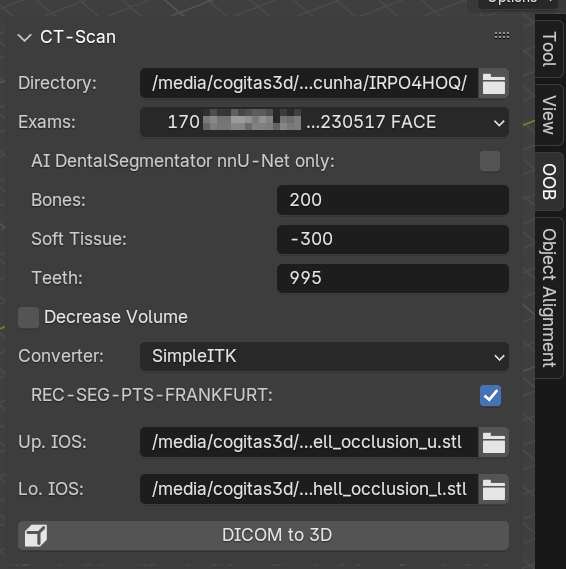
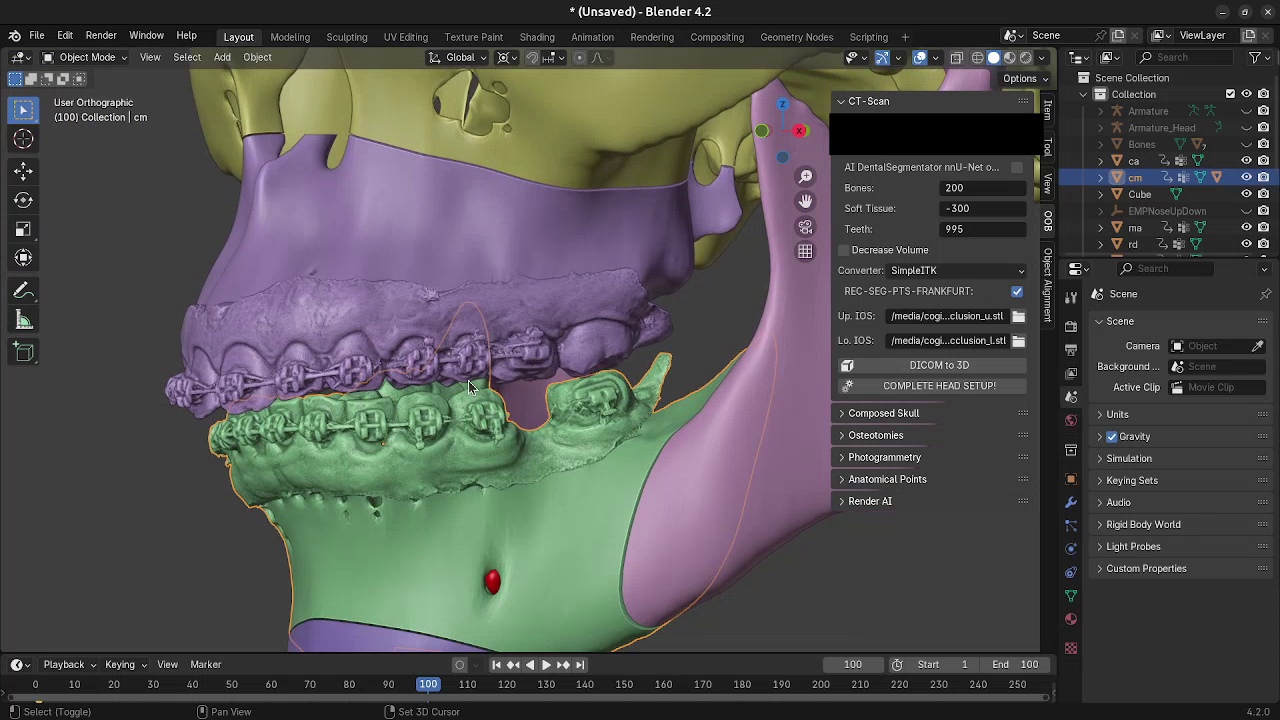
Fig. 18 Dados prontos para a reconstrução.
A diferença fundamental para ativar o sistema unificado é clicar no check button “REC-SEG-PTS-FRANKFURT”. Assim que a ação é efetuada, a interface se expande, apresentando dois campos de seleção dos arquivos STL das arcadas superior (Up. IOS) e inferior (Lo. IOS) (Fig. 18). A seleção é opcional, o sistema pode rodar sem que tais arquivos sejam setados, no entanto, é praxe que em um planejamento os escaneamentos intraorais (IOS) estejam presentes. Uma vez configurado, o botão “DICOM to 3D”, logo abaixo, pode ser pressionado, dando início ao processo.
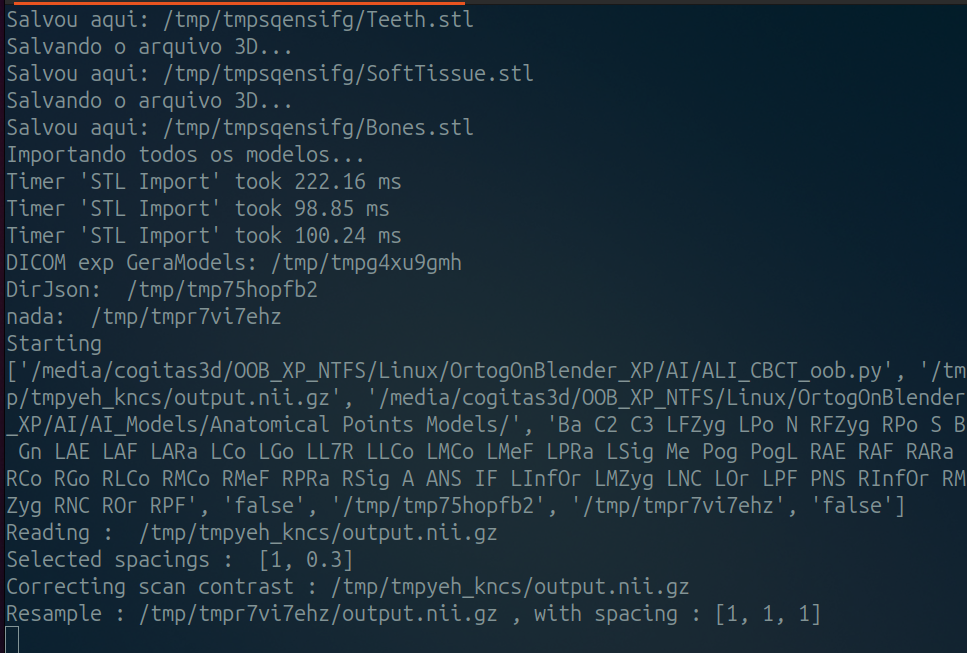
Fig. 19 Processo unificado rodando em background.
As etapas de trabalho passarão a rodar em background e podem ser companhadas pelo terminal (Fig. 19).
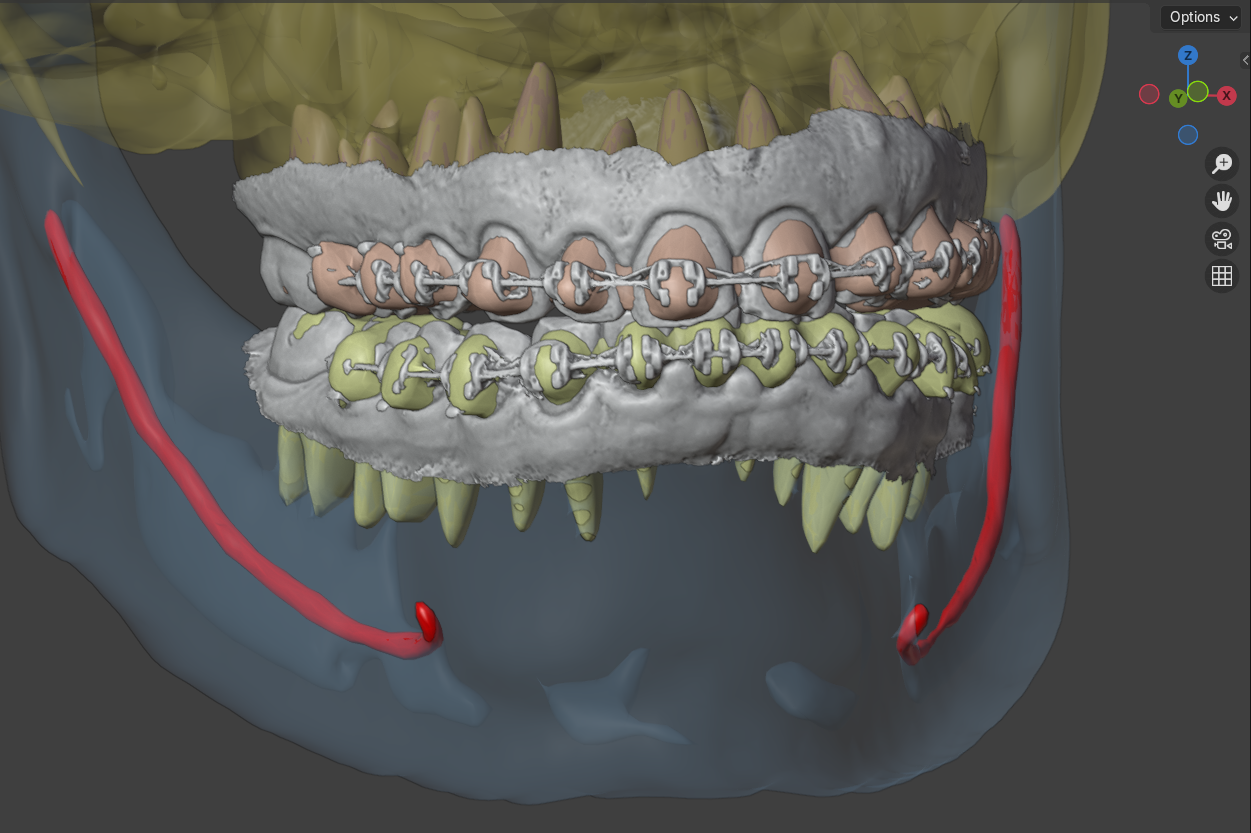
Fig. 20 Finalização bem-sucedida de todas as etapas do método unificado.
Após alguns minutos a interface é atualizada com o resultado do processo, resultando no caso do exemplo, na reconstrução de todos os tecidos (mole e ósseo), além da segmentação de regiões de interesse, bem como no alinhamento dos modelos dos dentes às segmentações das arcadas (Fig. 20). É importante deixar claro que a etapa final, ou seja, o alinhamento dos dentes nem sempre é bem sucedido como no exemplo desde material, sendo necessária uma etapa complementar de alinhamento, mas mesmo assim, a evolução em relação à versão anterior é significaticamente grande.
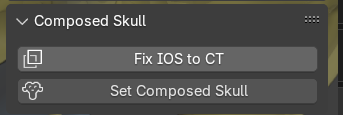
Fig. 21 Ajuste no alinhamento das arcadas.
Caso os modelos não estejam satisfatóriamente alinhados às arcadas, uma etapa complementar de ajuste pode ser feita através do botão “Fix IOS to CT” (Fig. 21). Como as estruturas estão nomeadas, o sistema se encarrega de proceder com os comandos, sem a necessidade do usuários selecionar nenhuma das malhas, bastando apenas ajustar uma melhor posição dos modelos para ajudar o algoritmo de alinhamento. Caso o alinhamento se estabeleça de modo satisfatório, o usuário poderá clicar em “Set Composed Skull”, para criar o crânio composto.
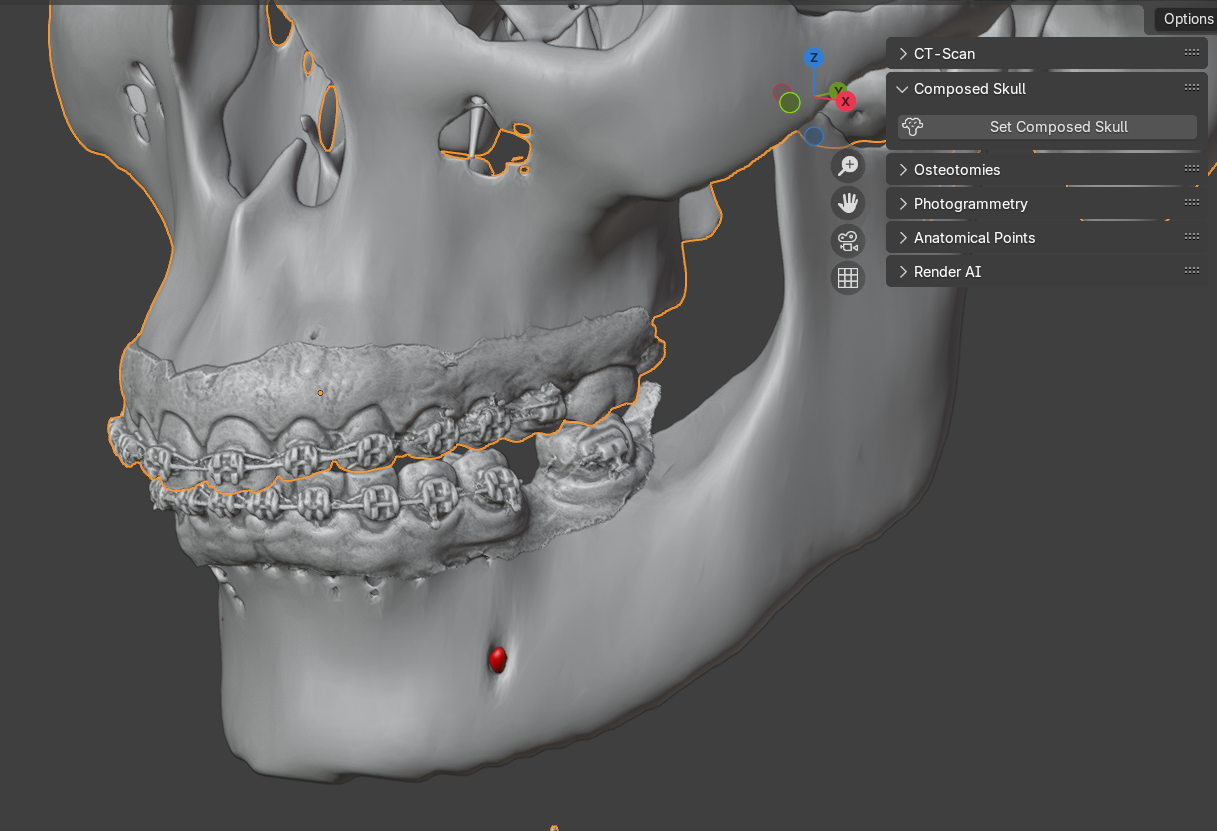
Fig. 22 Criação do crânio composto.
O sistema se encarregará de fundir os modelos com o crâno e a mandíbula, gerando o crânio composto em duas peças separadas, a “Composed_Skull” e a “Composed_Mandible” (Fig. 22).
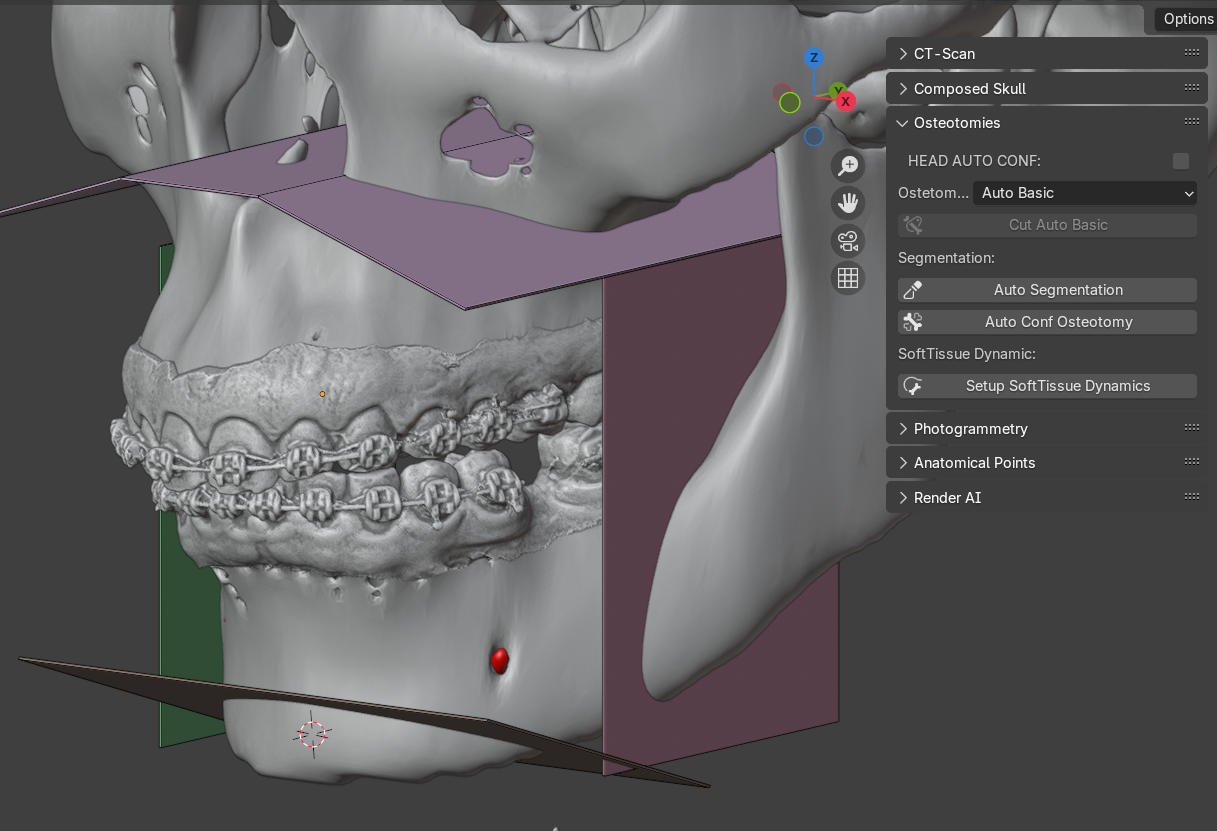
Fig. 23 Planos de corte criados.
Uma vez que o crânio composto esteja finalizado, é possível inserir os planos de corte na cena. Para isso é preciso expandir a seção “Osteotomies” e clicar em “Cut Auto Basic”, assim o sistema utilizará como referência os pontos anatômicos automaticamente posicionados nos ossos para distribuir os planos de corte (Fig. 23). Na imagem de exemplo o botão está desabilitado, pois acabou de ser usado, tornando desnecessário o comando, no entanto, há a possibilidade do usuário posicionar manualmente um por um dos planos de corte, bastando para isso trocar a opção de “Auto Basic” no menu logo acima, para “Manual Basic”.
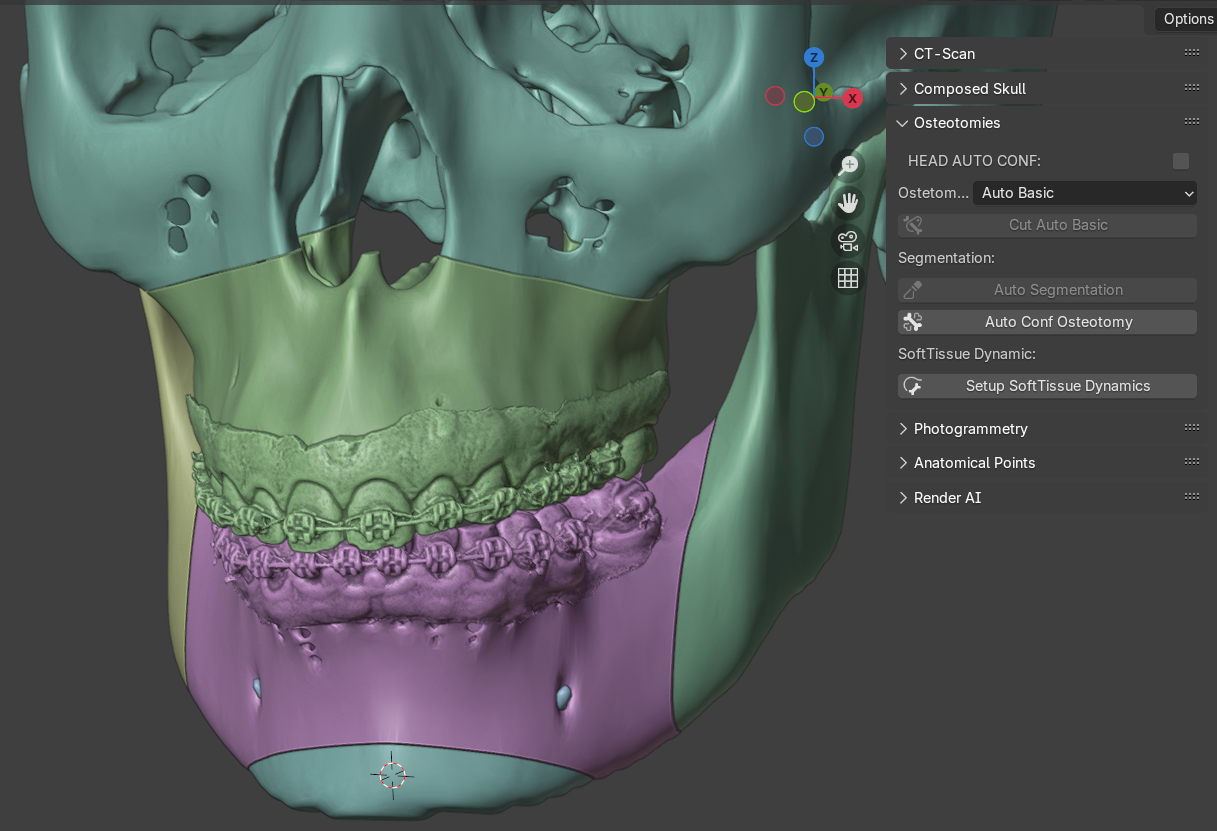
Fig. 24 Auto segmentação.
Apesar do sistema distribuir os planos de corte em locais coerentes com os objetivos da cirurgia ortognática, é imprescindível que o especialista ajuste as estruturas conforme as necessidades avaliadas por ele. Em seguida, basta clicar em “Auto Segmentation” para que as segmentações (osteotomias) se efetivem. O OOB_XP automaticamente troca o tipo de visualização de cores para random, de modo que o usuário avalie se o processo de corte foi bem sucedido (Fig. 24). Novamente, não é necesserário fazer nenhum tipo de seleção, pois o sistema o faz de modo autônomo. Apesar do OOB_XP fazer um a limpeza automática em malhas que não satisfaçam as estruturas esperadas para uma osteotomia, o usuário precisará checar se o número de ossos está correto e se tem algum resíduo indesejado. Sabendo-se que as osteotomias estão corretas, o usuário pode prosseguir com o próximo comando, que implica em clicar no botão “Auto Conf Osteotomy”.
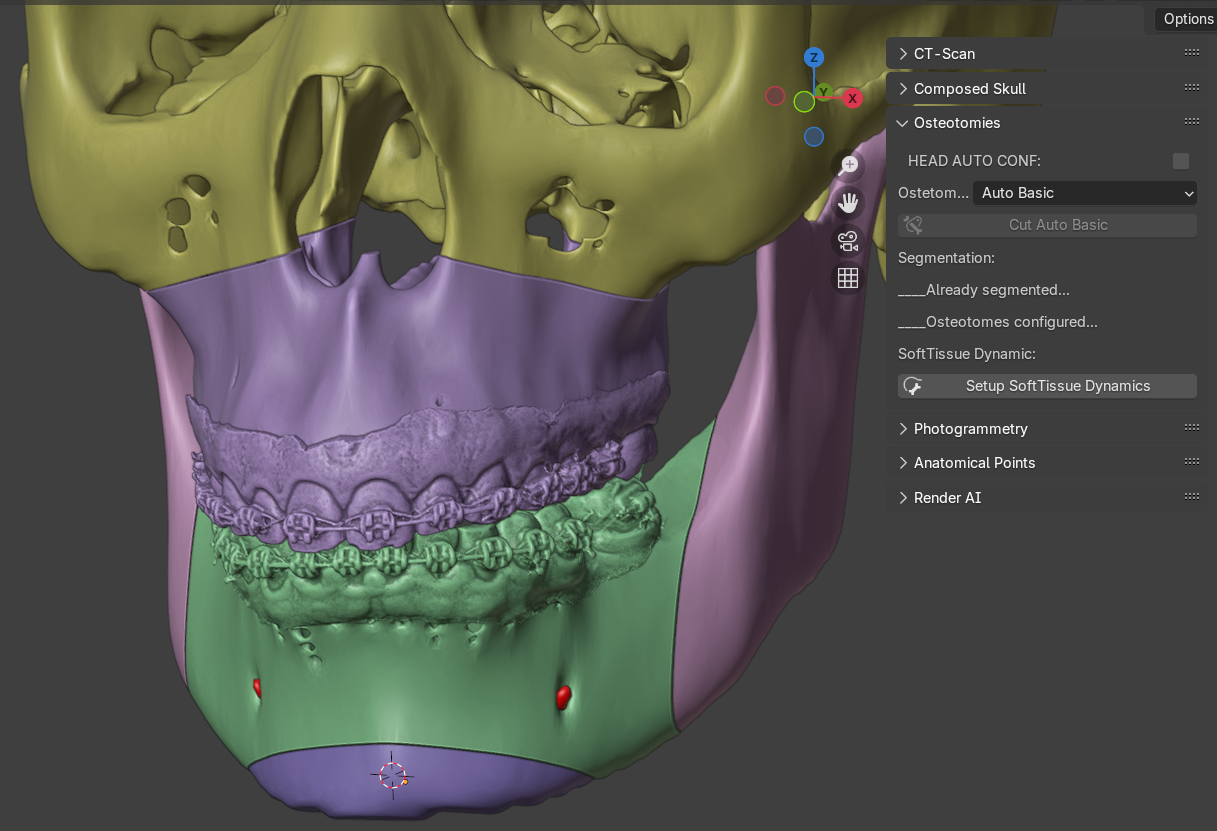
Fig. 25 Configuração das osteotomias.
O sistema se encarrega de fazer a configuração dos ossos, nomeando-os e criando a estrutura que será utilizada para a deformação do tecido mole (Fig. 25). Conforme os botões vão sendo utilizados e as condições satisfeitas, alguns deles são desabilitados ou sofrem uma alterçaão de estrutura, não sendo possível clicar novamente nos mesmos (a menos que se apaguem algumas estruturas ou se use o comando de voltar). Estando tudo ajustado, o usuário pode clicar em “Setup SoftTissue Dynamics” para configurar a deformação do tecido mole.
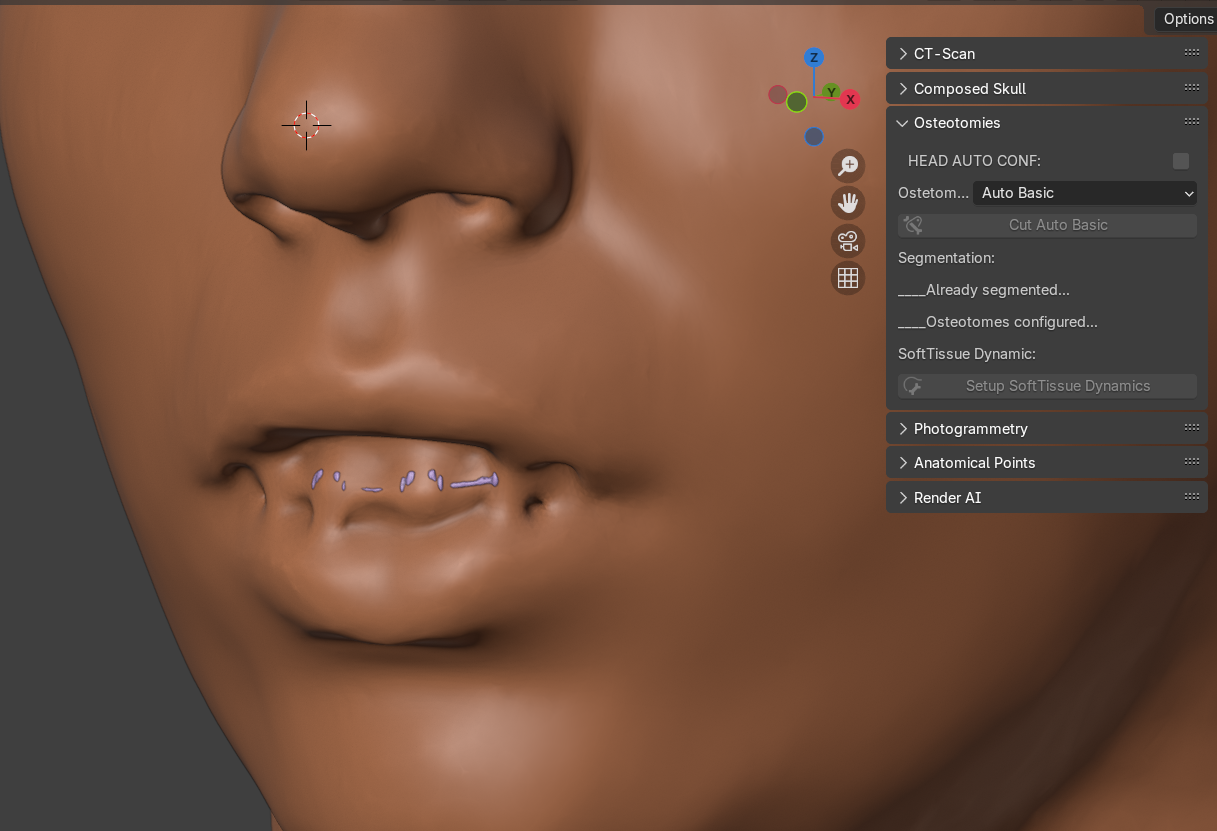
Fig. 26 SoftTissue dynamic efetuado.
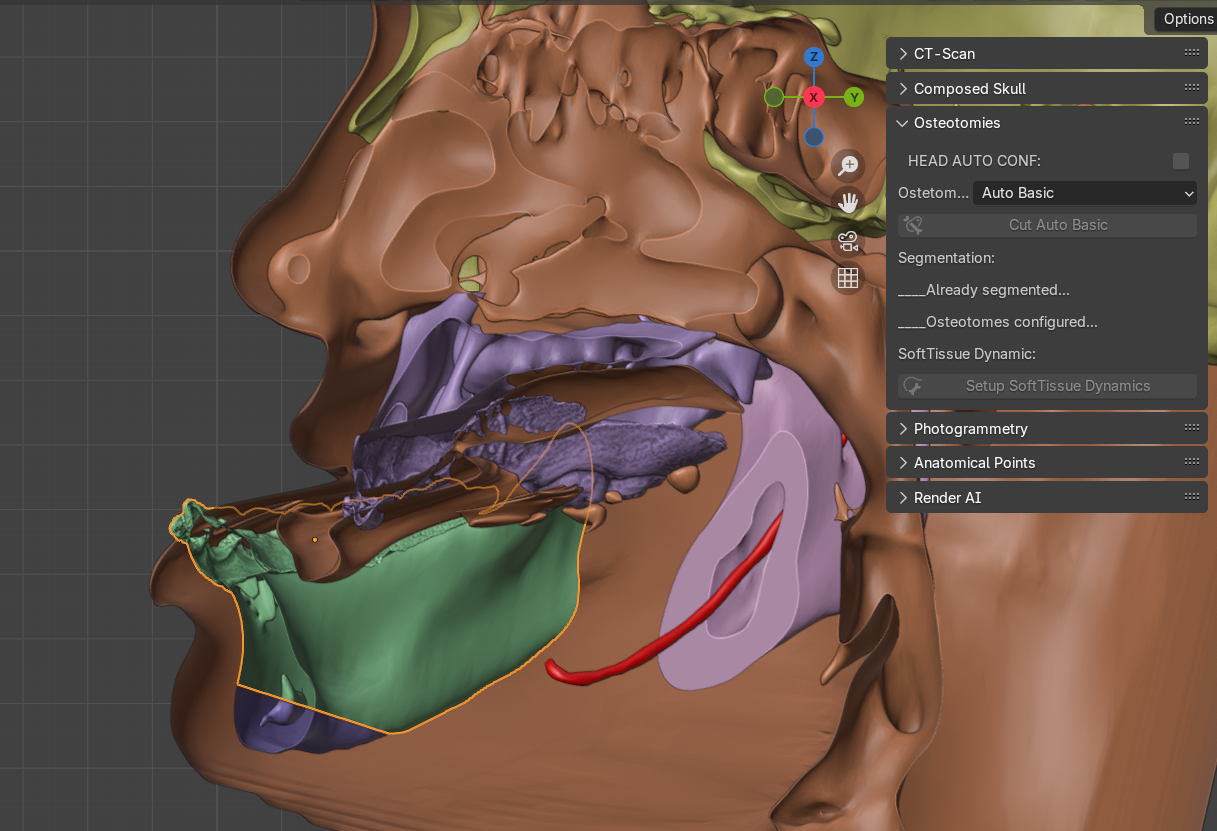
Fig. 27 Teste de deformação.
Novamente não é necessário fazer nenhum tipo de seleção, pois o OOB_XP se encarrega de projetar as áreas de deformação conforme as osteotomias (Fig. 26), além de fazer correções nos limites dos lábios, o que não estava disponível na versão anterior do add-on. Não é necessário o usuário selecionar a malha do tecido mole, tampouco torná-la visível, pois o add-on faz isso automaticamente (Fig. 27).
Detalhes Técnicos e Testes
Estrutura dos Comandos

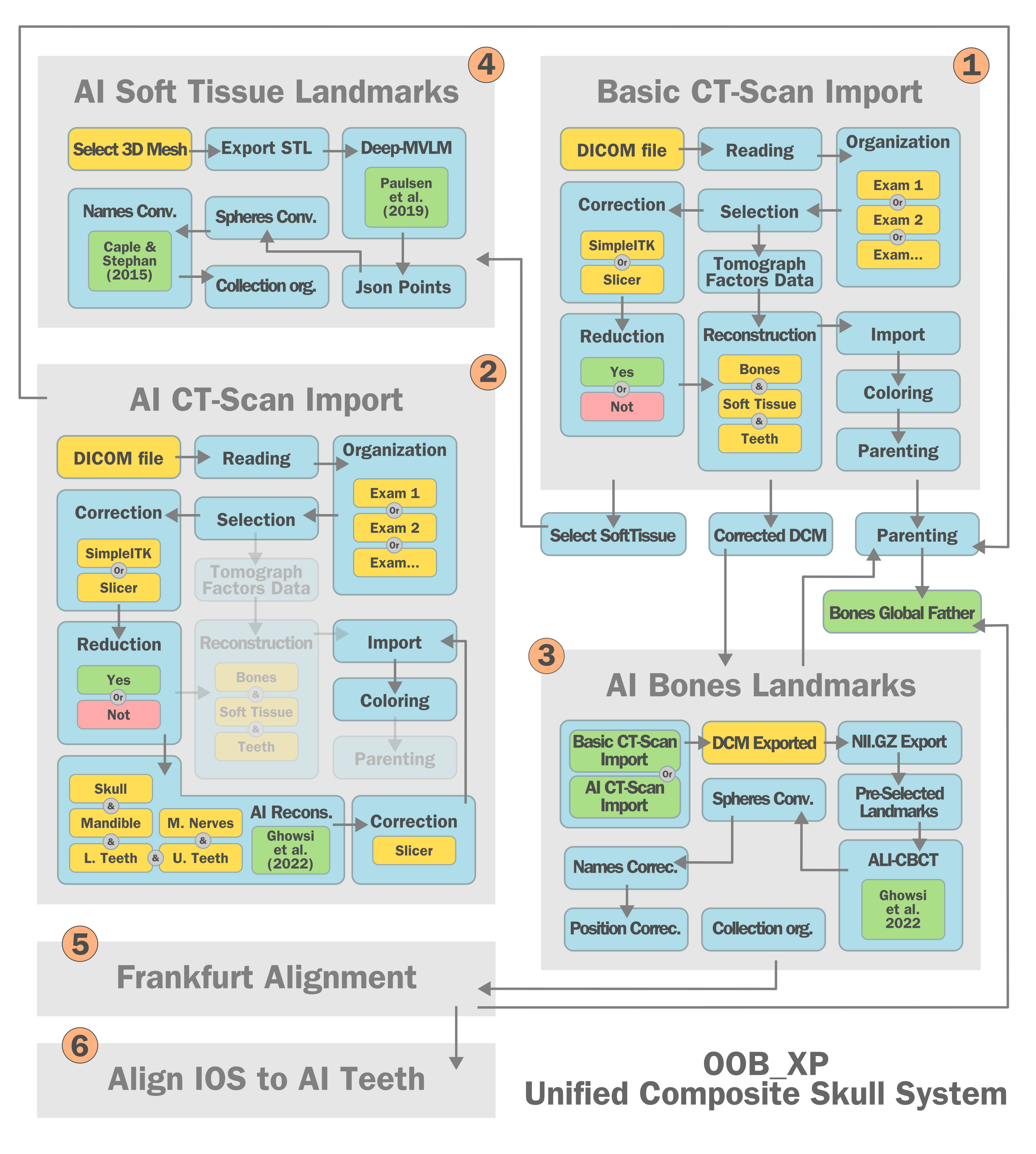
Fig. 28 Esquema do sistema unificado para a criação do crânio composto.
O sistema unificado para a criação do crânio composto é uma evolução de abordagens que vêm sendo testadas e utilizadas no OrtogOnBlender ao longo dos anos, com a incrementação de novas ferramentas baseadas em inteligência artificial. O esquema simplificado (Fig. 28) ilustra como a estrutura funciona:
Basic CT-Scan Import: Depois do usuário selecionar o exame que se pretende trabalhar, a reconstrução básica dos ossos, tecido mole e dentes é efetuada. Tal passo é necessários, pois é dali que sairá a malha do tecido mole (SoftTissue) usada na deformação cirúrgica, o crânio (Bones) que será o pai de todas as outras malhas e os dentes (Teeth) que fornecerão dados complementares da estrutura dos dentes e eventualmente de algum artefato de metal presente na cabeça. Essa etapa foi descrita em [B_Moraes_et_al_2024].
AI CT-Scan Import: Em seguida há uma etapa de segmentação do crânio, da mandíbula, da arcada superior, arcada inferior e dos nervos mentuais. Essa fase simplifica muito o trabalho, pois ao usuário será poupada a etapa, muitas vezes enfadonha, de segmentação manual. As malhas resultantes são parenteadas como filhas do crânio principal (Bones). Essa ferramenta foi descrita em [B_Moraes_et_al_2024b]. Leitura complementar [B_Dot_et_al_2024] [B_Isensee_et_al_2020].
AI Bones Landmarks: Com as estruturas reconstruídas, outra ferramenta baseada em inteligência artificial foi utilizada para econtrar uma série de pontos anatômicos no tecido ósseo. Tal ferramenta já havia sido estudada anteriormente e está descrita em [B_Moraes_et_al_2024c]. Leitura complementar: [B_Gillot_et_al_2023].
AI Soft Tissue Landmarks: Um importante passo foi adicionado no processo, este utiliza a inteligência artificial para distribuir uma série de pontos ao longo da malha da face. A ferramenta está descrita em [B_Paulsen_et_al_2019] e passou por algumas adaptações para se adequar à interface gráfica do OOB_XP, como a conversão dos números dos pontos em nomes das regiões anatômicas, tomando como referência o trabalho de [B_Caple_and_Stephan_2015].
Frankfurt Alignment: Utilizando os dados dos pontos anatômicos, além de alguns parâmetros para fortalecer a coerência estrutural, o crânio principal é alinhado ao plano horizontal de Frankfurt e uma vez que os demais elementos da cena estão parenteados a ele, todos seguem a rotação.
Align IOS to AI Teeth: Uma série de comandos implementados pelos autores posiciona os modelos dos dentes (IOS) próximos aos dentes extraídos pelas ferramentas de AI e refinam o alinhamento com ICP, a partir do trabalho de Moore et al. (https://github.com/patmo141/object_alignment).
Todas as ferramentas receberam camadas adicionais de ajustes para que o resultado fosse unificado em apenas um espaço e interface gráfica. O OOB_XP complementou as etapas com mecanismos anti-erro, de modo que o usuário tenha o mínimo de interação e o máximo de acerto ao utilizar a solução.
Posteriormente foram portadas outras ferramentas à interface do OOB_XP, como o posicionamento automático de planos de cortes (osteotomias), o próprio corte digital (boolean), a configuração automática das osteotomias e finalmente a configuração da deformação do tecido mole. Tais ferramentas já faziam parte das versões anteriores do OOB, bem como já haviam passado por comparações do estilo planejamento digital vs. pós cirúrgico real [B_Cunha_et_al_2020] [B_Lobo_et_al_2022], resultando em significativa compatibilidade. Tais ferramentas passaram por um processo de melhoramento funcional, graças à experiência e feedback dos usuários, bem como com o incremento da AI no código.
Attention
Durante o desenvolvimento da ferramenta, foi criado um botão com uma cadeia de comandos, envolvendo todos os processos descritos acima, bastando ao usuário indicar o local dos arquivos DICOM, qual exame seria utilizado e a informação dos IOS superior e inferior. Com essas informações o OOB_XP foi capaz de, sem a intervenção do usuário, proceder autonomamente desde a reconstrução da tomografia, segmentação, alinhamento em Frankfurt, passando pelo alinhamento dos IOS às segmentações dos arcos, fundindo as estruturas para a criação do crânio composto, posicionando os planos de corte, efetuado as osteotomias, configurando as osteotomias e a deformação do tecido mole. Um vídeo timelapse foi gravado, ilustrando todo o processo: https://youtu.be/RRLIiqwSMKk.
Para assistir o vídeo clique sobre o thumbnail acima ou no link da caixa informativa.
Warning
Embora seja possível proceder de modo automático com todos os comandos descritos acima, os desenvolvedores não recomendam que isso seja efetuado durante o processo de planejamento cirúrgico, uma vez que a participação do cirúrgião nas decisões é essencial para uma simulação bem sucedida, reforçando a segurança do processo.
Testes
Seguindo o padrão de testes efetuados a cada nova ferramenta do OOB, além das 404 tomografias reconstruídas no conjunto 1 (Basic CT-Scan Import) e das 151 na sessão 2 (AI CT-Scan Import), também procedeu-se com 115 para o sistema de 3 (AI Bones Landmarks), 101 para o sistema 4 (AI Soft Tissue Landmarks), e 101 para o sistema 5 (Frankfurt Align). Também foram efetuados 120 testes para o sistema unificado, com exceção do alinhamento dos dentes, além de 12 testes em três sistemas operacionais diferentes (12x3=36) com o sistema unificado completo. Ao todo foram efetuados 1028 testes, visando analisar aspectos específicos do funcionamento das ferramentas em fases diferentes.
Configuração da máquina: Processador Intel Core I9 9900K 3.6 GHZ/16M; 64 GB de memória RAM; GPU GeForce 8 GB GDDR6 256-bit RTX 2070; Placa mãe Gigabyte 1151 Z390; SSD SATA III 960 GB 2.5”; SSD SATA III 480 GB 2.5”; Water Cooler Masterliquid 240V; Linux Ubuntu 24.04 e Windows 11 (armazenados em mídias distintas).
Nos testes básicos chegou-se a uma taxa de 100% de sucesso nas reconstruções, desde que resolvidas as incompatibilidades das tomografias. O mesmo nível de sucesso se deu na segmentação por AI, já os testes com o posicionamento dos pontos, tanto nos ossos quanto nos tecidos moles, serviram para avaliar quais pontos eram mais constantes, de modo a utilizá-los para alinhamentos, poisicionamento de estruturas e limitação de áreas deformáveis. O teste do sistema unificado sem o alinhamento dos dentes serviu para avaliar se os pontos selecionados para o alinhamento em Frankfurt funcionariam em casos reais, resultanto em uma taxa de sucesso de 93,81% e uma média de tempo de 11 minutos e 10 segundos para cada processamento. Curiosamente, dos erros de alinhamento registrados, dois deles se deram por deformações ósseas nos pacientes e 4 ocorreram em tomografias médicas, indicando um melhor funcionamento em DICOMs advindos de CBCT.
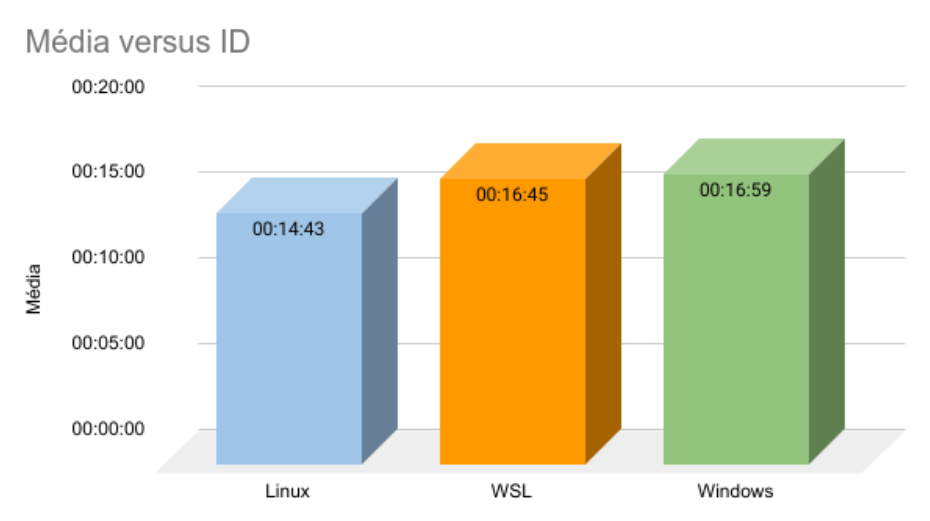
Fig. 29 Comparação entre sistemas diferentes.
Foram escolhidos os dados aleatórios de 12 pacientes anonimizados, com os quais foram rodados todos os passos do Sistema Unificado para a Criação de um Crânio Composto no OrtogOnBlender XP. Esse conjunto de 12 pacientes foi testado em três sistemas distindos, no Ubuntu Linux 24.04 rodando nativamente, no Windows 11 rodando nativamente e no Linux Ubuntu 24,04 rodando dentro do Windows 11 (WSL 2). Apesar de algumas pequenas diferenças estruturais e de adptações efetuadas para a compatibilização em cada sistema operacional, não houve grande diferença de resultados de tempo e aparência (Fig. 28). O Linux se saiu ligeiramente melhor nos dois quesitos.
Conclusão
Em face aos resultados, alicerçados em tomografias de pessoas reais, em sua maioria com deformações faciais, o Sistema Unificado para a Criação de um Crânio Composto no OrtogOnBlender XP se mostrou suficientemente robusto e pronto para ser compartilhado com os seus usuários.
Agradecimentos
Ao Dr. William Sanchis, por ceder a tomografia computadorizada utilizada neste estudo. Um agradecimento especial a todos aqueles que enviaram tomografias computadorizadas, formando um grupo de arquivos importantíssimo para testar o uso do sistema em situações práticas.
Referências Bibliográficas
Caple J, and Stephan C, N. A standardized nomenclature for craniofacial and facial anthropometry. 2015. URL: http://dx.doi.org/10.1007/s00414-015-1292-1, doi:10.1007/s00414-015-1292-1.
Cunha, H. S., da Costa Moraes, C. A., de Faria Valle Dornelles, R., & da Rosa, E. L. S. (2020). Accuracy of three-dimensional virtual simulation of the soft tissues of the face in OrtogOnBlender for correction of class II dentofacial deformities: an uncontrolled experimental case-series study. In Oral and Maxillofacial Surgery (Vol. 25, Issue 3, pp. 319–335). Springer Science and Business Media LLC. https://doi.org/10.1007/s10006-020-00920-0
Dot, G., Chaurasia, A., Dubois, G., Savoldelli, C., Haghighat, S., Azimian, S., Taramsari, A. R., Sivaramakrishnan, G., Issa, J., Dubey, A., Schouman, T., & Gajny, L. (2024). DentalSegmentator: Robust open source deep learning-based CT and CBCT image segmentation. In Journal of Dentistry (Vol. 147, p. 105130). Elsevier BV. https://doi.org/10.1016/j.jdent.2024.105130
Gillot, M., Miranda, F., Baquero, B., Ruellas, A., Gurgel, M., Al Turkestani, N., … Prieto, J. C. (2023). Automatic landmark identification in Cone‐beam computed tomography. Orthodontics & Craniofacial Research, 26(4), 560–567. doi:10.1111/ocr.12642
Isensee, F., Jaeger, P. F., Kohl, S. A. A., Petersen, J., & Maier-Hein, K. H. (2020). nnU-Net: a self-configuring method for deep learning-based biomedical image segmentation. In Nature Methods (Vol. 18, Issue 2, pp. 203–211). Springer Science and Business Media LLC. https://doi.org/10.1038/s41592-020-01008-z
Lobo, F., Filho, L. I., Sigua-Rodriguez, E. A., da Silva, B. G., Tolentino, E. de S., Borges, Y. M., da Silva, M. C., Tonin, R. H., & Iwaki, L. C. V. (2022). Evaluation of ortogonblender software bone movement tools in bimaxillary orthognatic surgeries performed in dolphin software. In Journal of Stomatology, Oral and Maxillofacial Surgery (Vol. 123, Issue 4, pp. 417–421). Elsevier BV. https://doi.org/10.1016/j.jormas.2021.10.001
Moraes, C., Dakir, I., Startek, B., Dornelles, R., & Rosa, E. da. (2024). Ferramenta de Importação de Tomografia Computadorizada no OrtogOnBlender XP. figshare. https://doi.org/10.6084/M9.FIGSHARE.27648876. https://ortogonline.com/doc/pt_br/OrtogOnLineMag/10/CT_Scan.html
Moraes, C., Dakir, I., Startek, B., Dornelles, R., & Rosa, E. da. (2024). Segmentação de Tomografias Computadorizadas por IA no OrtogOnBlender XP. figshare. https://doi.org/10.6084/M9.FIGSHARE.27761970. https://ortogonline.com/doc/pt_br/OrtogOnLineMag/10/AI.html
Moraes, C., Schreiner, T., Startek, B., & Beaini, T. L. (2024). Alinhamento em Frankfurt, Colocação de Pontos Anatômicos e Segmentação do Crânio por Inteligência Artificial no OrtogOnBlender. figshare. https://doi.org/10.6084/M9.FIGSHARE.25140020. https://ortogonline.com/doc/pt_br/OrtogOnLineMag/8/AI.html
Paulsen, R. R., Juhl, K. A., Haspang, T. M., Hansen, T., Ganz, M., & Einarsson, G. (2019). Multi-view Consensus CNN for 3D Facial Landmark Placement. In Lecture Notes in Computer Science (pp. 706–719). Springer International Publishing. https://doi.org/10.1007/978-3-030-20887-5_44