Segmentação de Tomografias Computadorizadas por IA no OrtogOnBlender XP
Atenção
Este material utiliza a seguinte licença Creative Commons: Atribuição 4.0 Internacional (CC-BY 4.0).
Atenção
Caso encontre algum erro no texto, sinta-se à vontade para informá-lo aos autores, o contato pode ser feito via redes sociais acadêmicas informadas no início do capítulo.
Introdução
O presente capítulo apresenta a nova ferramenta de reconstrução e segmentação de tomografias computadorizadas (CT-Scan e CBCT) utilizando inteligência artificial (IA) baseada no DentalSegmentator e uuN-Net [F_Dot_et_al_2024] [F_Isensee_et_al_2020]. A mesma está sendo desenvolvida para o OrtogOnBlender XP (OOB_XP), uma versão baseada no Blender 4.2 LTS, inicialmente para o Linux Ubuntu 24.04 LTS e posteriormente para o Windows e o MacOS. Esforços passados implementaram algumas ferramentas de IA na versão release do OrtogOnBlender (OOB) [F_Moraes_et_al_2020], tanto para a segmentação dos dentes em modelos escaneados [F_Moraes_and_Dakir_2021] quando para a segmentação dos ossos faciais, localização de pontos faciais e alinhamento no plano de Frankfurt [F_Moraes_et_al_2024].
Como Funciona
O funcionamento segue praticamente o mesmo passo-a-passo já abordado em [F_Moraes_et_al_2024b], com algumas pequenas diferenças.
Fig. 71 Seletor de dirtetório DICOM.
A seção de importação de tomogrfias computadorizadas está na aba OOB, na lateral da 3D View. O primeiro passo é selecionar o diretório onde se encontra a sequência de arquivos ou arquivo único DICOM, clicando no ícone de diretório à direita (Fig. 71).
Fig. 72 Momento da seleção do diretório.
Uma janela de seleção será aberta, na qual o usuário indicará o local onde se encontram os arquivos DICOM. É recomendado que o diretório seja a raiz onde se encontram os arquivos DICOM ou mesmo os diretórios com arquivos DICOM, buscando evitar estruturas anteriores, como por exemplo, a raiz da mídia onde está o exame, pois geralmente contam com muitos diretórios sem arquivos DICOM, o que elevaria o tempo de busca e potencialmente dar magem a geração de erros. Uma vez que o diretório é indicado, basta clicar em “Accept” (Fig. 72).
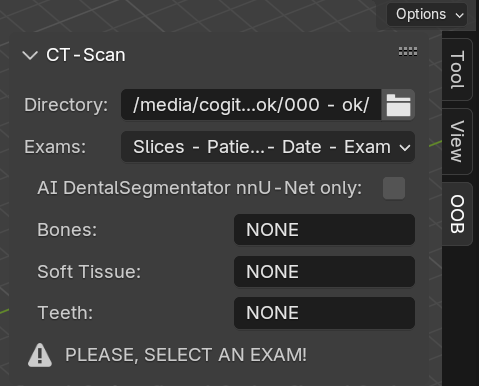
Fig. 73 Interface básica carregada.
Assim que o diretório é selecionado, leva um certo tempo até que os exames DICOM sejam organizados. Uma vez que o processo é efetuado, a interface básica é apresentada (Fig. 73), no entanto, para utilizar a ferramenta de IA, um passo adiconal é necessário.
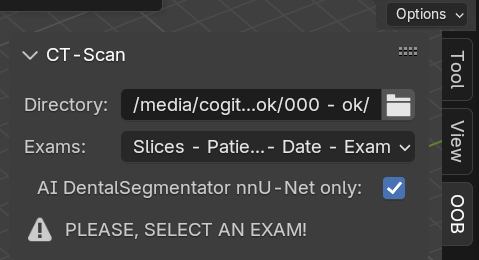
Fig. 74 Habilitando a IA.
Tal passo implica em habilitar o checkbox de “AI DentalSegmentator nnU-Net only”, o que tornará a interface mais enxuta (Fig. 74), uma vez que o sistema não necessita do preenchimento dos fatores relacionados aos tecidos que se pretende reconstruir. Outrossim, o usuário precisa selecionar o exame que pretende reconstruir, como indica a mensagem de atenção “PLEASE, SELECT AN EXAM!”.
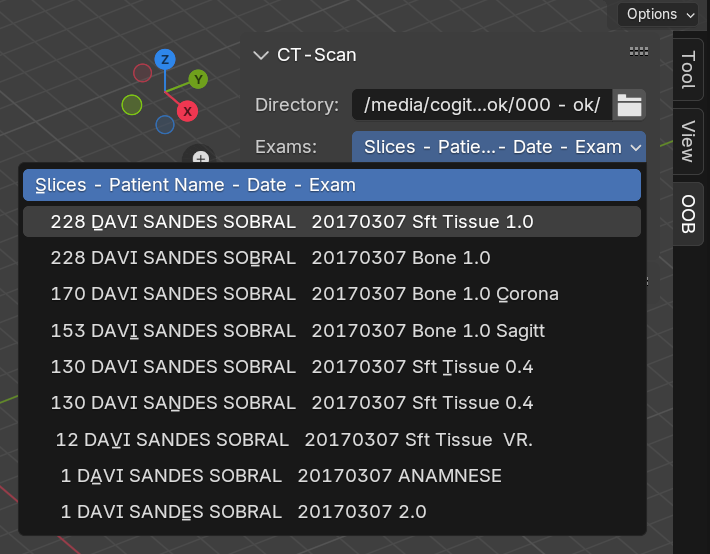
Fig. 75 Seleção do exame desejado.
A seleção é feita, clicando no menu “Exams”, e selecionado aquele desejado. A listagem é ordenada pelo número de fatias do exame e as informações em ordem horizontal são as seguintes: 1) Número de fatias, 2) Nome do Paciente, 3) Data do exame e 4) Descrição do exame (Fig. 75).
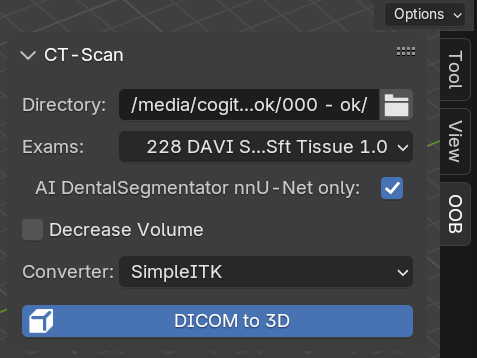
Fig. 76 Acionando o processo de reconstrução 3D.
Selecionado o exame, o passo seguinte implica em clicar no botão “DICOM to 3D” para recontruir as malhas correspondentes aos tecidos pré-configurados (Fig. 76).
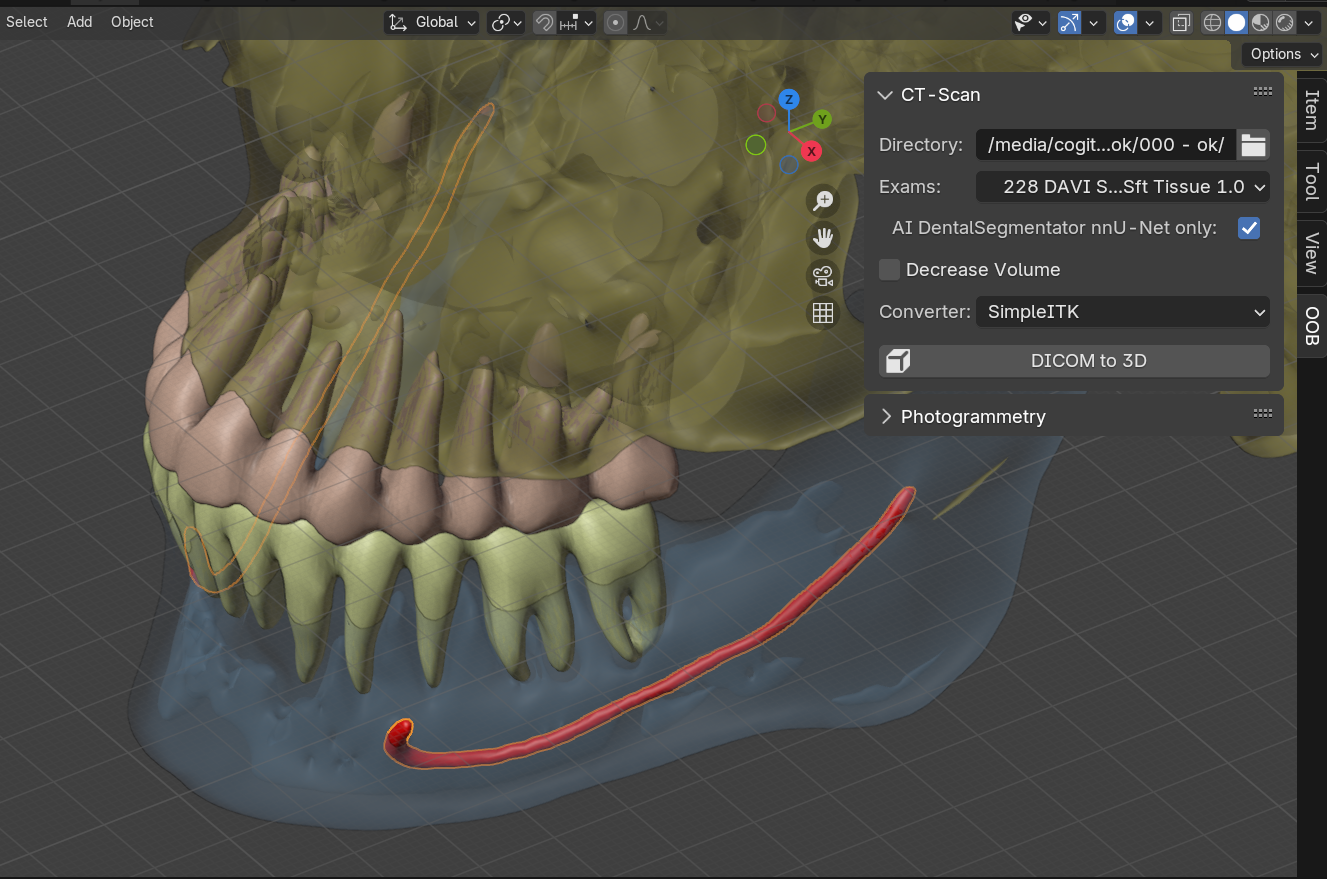
Fig. 77 Malhas reconstruídas por IA.
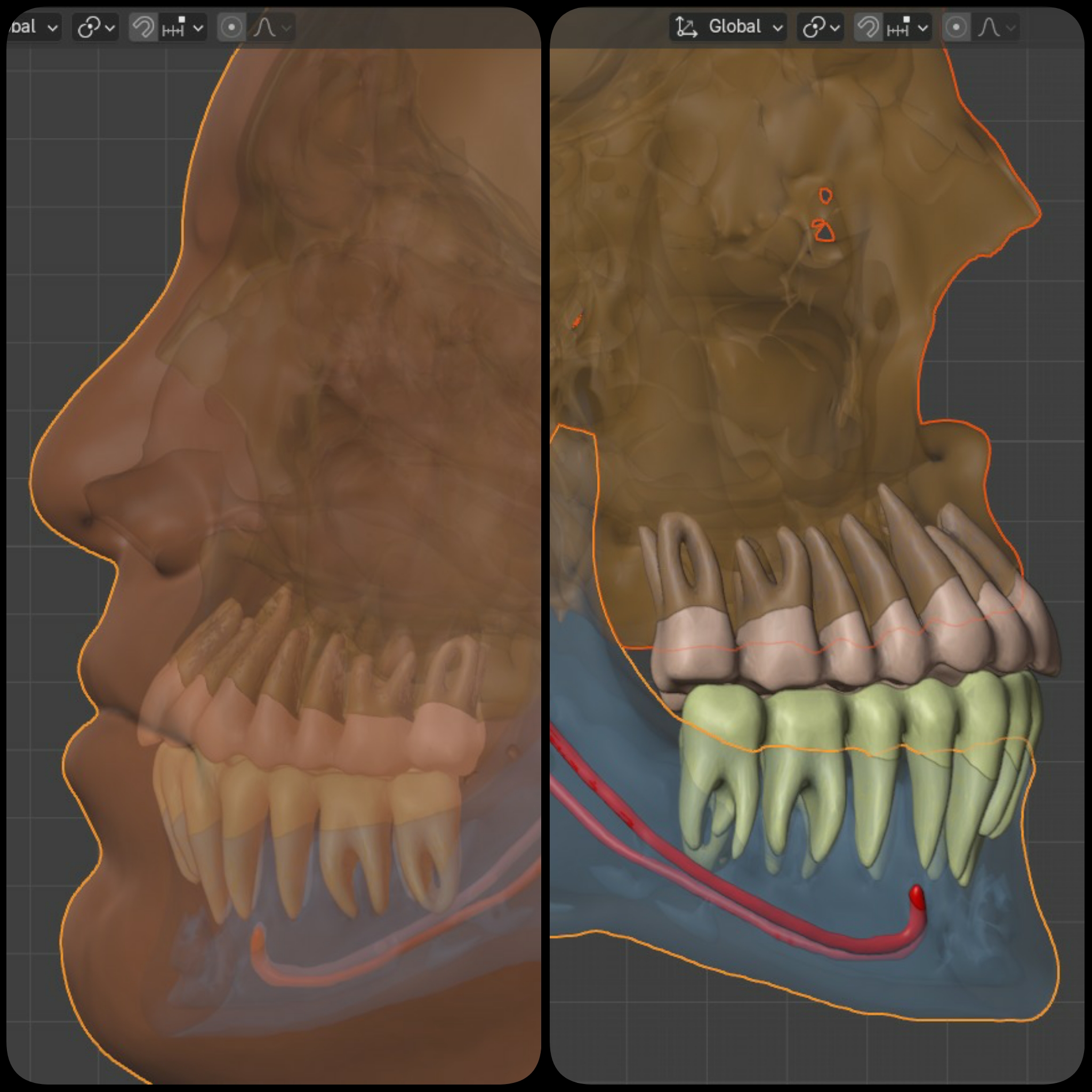
Assim que o processo de reconstrução é finalizado, as malhas correspondentes aos tecidos são importadas na cena, recebendo tratamento de cor e opacidade, para uma apresentação que propicie uma avaliação inicial de todos os modelos esperados: crânio (AI_Skull), mandíbula (AI_Mandible), dentes da arcada superior (AI_Teeth_Upper), dentes da arcada inferior (AI_Teeth_Lower) e nervos mentouais (AI_nerves) (Fig. 77).
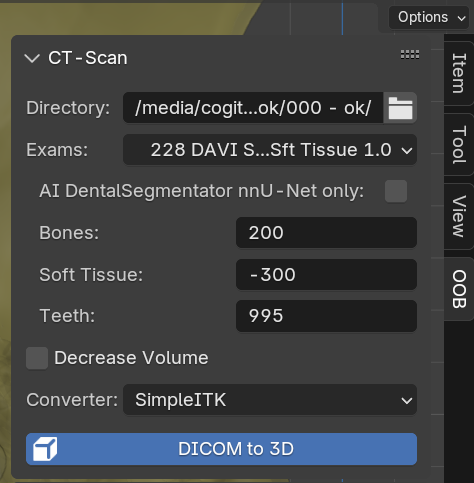
Fig. 78 Desabilitando a IA.
Os usuários do OOB geralmente necessitam de informações do tecido mole e como a ferramenta de IA não os reconstrói, uma solução prática e rápida envolve desabilitar o checkbox da IA, o que fará com que a interface expanda e apresente os dados dos fatores de reconstrução dos ossos, tecido mole e dentes, colhidos durante a organização dos exames. Para reconstruir as estruturas basta clicar em “DICOM to 3D” (Fig. 78).
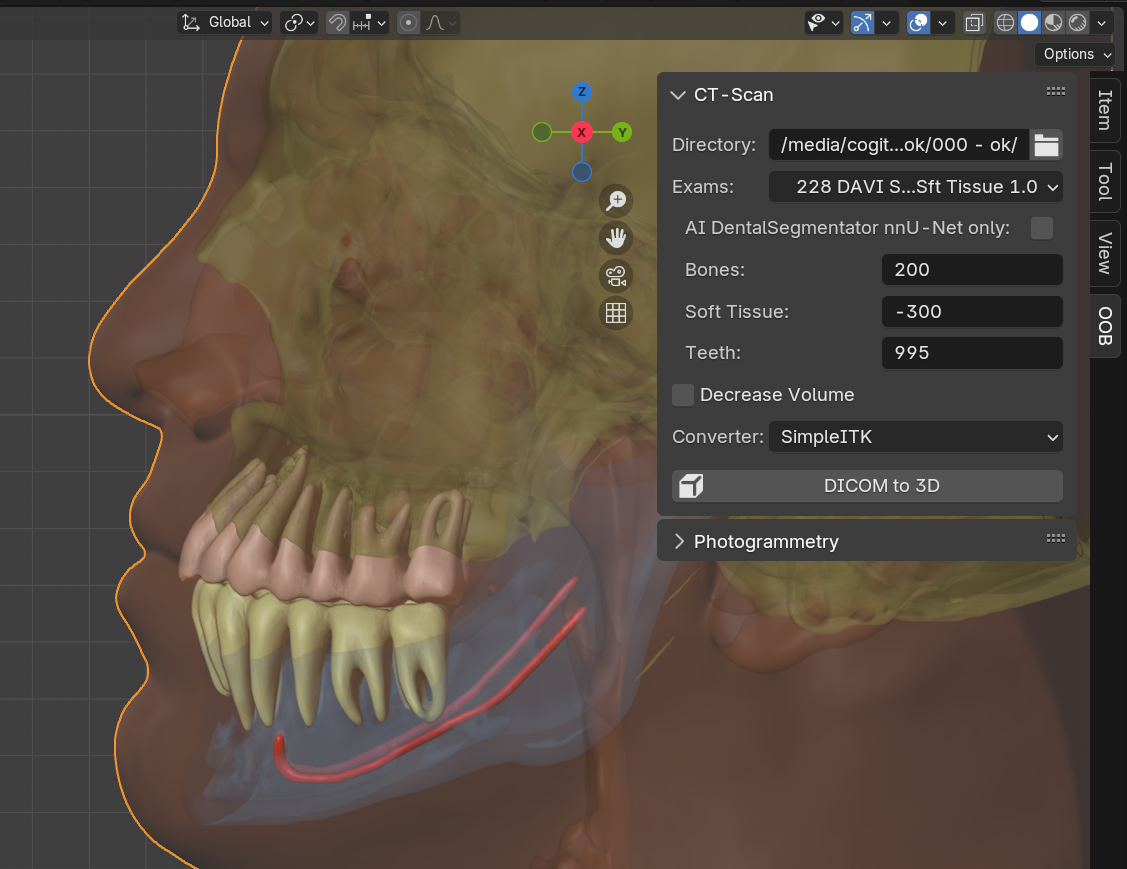
Fig. 79 Tecido mole adiconado na cena, com ajuste da transparência.
No exemplo, a malha dos ossos e dos dentes foram ocultas e a malha do tecido mole recebeu um ajuste na sua transparência para que os elementos da reconstrução anterior fossem visualizados (Fig. 79), algo trivial para um usuário médio de OOB. O ponto de interesse aqui é ilustrar que a importação das malhas é efetuada de modo que, mesmo se tratando de abordagens diferentes, a posição das estruturas é feita no mesmo espaço, permitindo que o usuário desfreute de todas as opções de reconstrução, para complementar os dados, caso seja necessário (e em muitos casos, de fato o é).
Características Técnicas e Testes
Estrutura do Script
A programação das ferramentas IA no OOB_XP foi inserida em um sistema já existente e legado de reconstrução de tomografias computadorizadas, permitindo que abordagens diferentes gerem estruturas que dividem o mesmo espaço final. Apesar das ferramentas DentalSegmentator e uuN-Net serem suficientemente robustas no meio para o qual foram originalmente criadas, ou seja, para a interface do Slicer (https://www.slicer.org/), ao portá-las para a interface do Blender (https://www.blender.org/) alguns desafios são adiconados no processo.
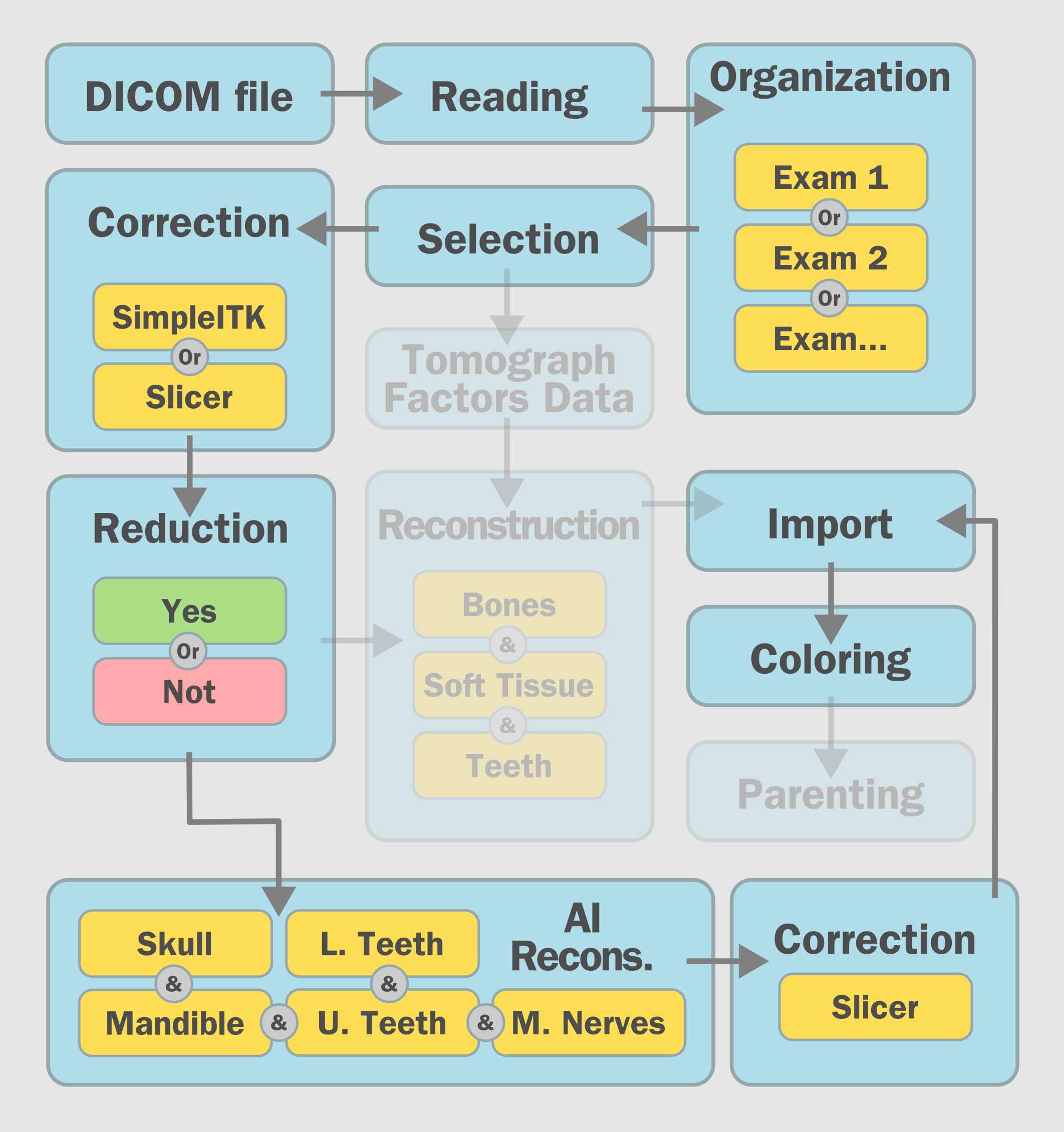
Fig. 80 Esquema de funcionamento da reconstrução por IA no OOB_XP.
O papel do OOB_XP é justamente contornar eventuais problemas e proporcionar ao usuário uma abordagem simples e objetiva, trabalhando no background para que as malhas sejam importadas sem maiores problemas. Caso o usuário encontre alguma incompatibilidade com a biblioteca SimpleITK, por exemplo, ele por optar por utlizar a solução baseada em linha de comando do Slicer. Nos testes iniciais, os scripts baseados em bibliotecas Python não estavam conseguindo reconstuir o arquivo NII.GZ resultante da segmentação por IA, então optou-se por corrigir essa incompatibilidade convertendo o arquivo final em DICOM, o que propiciou o controle necessário para importar modelos na mesma posição espacial daqueles efetuados pelas ferramentas clássicas do OOB. O esquema de funcionamento geral pode ser visto na imagem ilustrativa (Fig. 80), o que denota uma convergência satisfatória e de mantenimento acessível por parte dos desenvolvedores.
Testes com CT-Scans e CBCTs
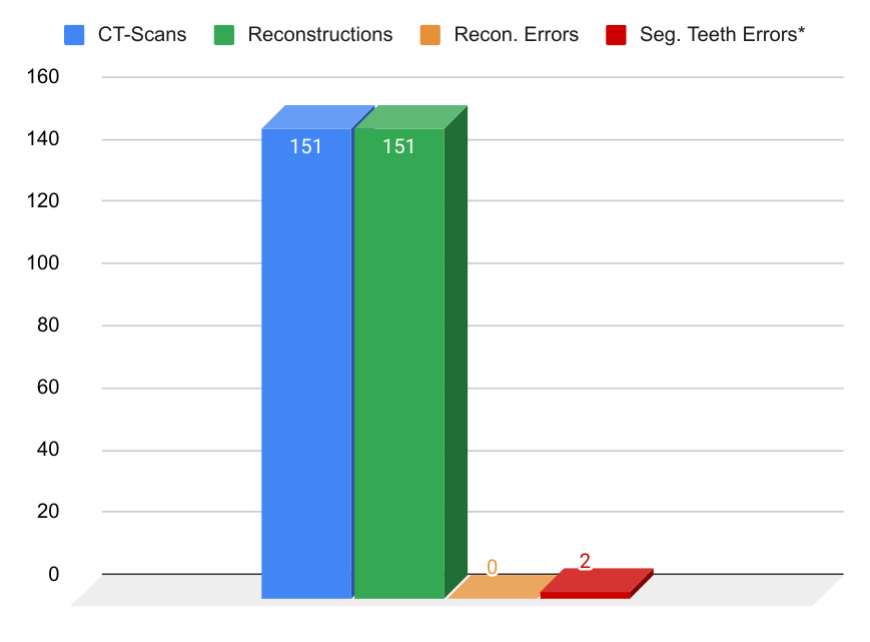
Fig. 81 Resultado das reconstruções, sendo 151 tomografias computadorizadas utilizadas (CT-Scan e CBCT), delas todas as 151 foram reconstruídas com a solução sitk+slicer+GUI+DentalSegmentator (Reconstruction), logo, nenhuma delas apresentou erro (Recon. Erros). * Apesar de duas tomografias terem apresentado erro de segmentação, uma delas de tratava de um indivíduo desdentado contendo apenas próteses inferiores e outro tratou-se de um indivíduo com a dentição inferior, mas desdentado na arcada superior, sendo assim, dependendo da abordagem pode ser considerado apenas um erro nos 151 levantamentos.
O OOB é um addon desenvolvido para especiaistas que trabalham com planejamento cirúrgico e ao longo dos anos, desde que foi lançado em 2017, uma série de artigos foram publicados, demonstrando a sua robustez nos mais variados campos, como cirurgia ortognática [F_Cunha_et_al_2020] [F_Lobo_et_al_2022], rinoplastia [F_Sobral_et_al_2021], fraturas mandibulares [F_Facanha_et_al_2021], expansores mandibulares de bebês [F_Duarte_et_al_2023], documentação urológica [F_Nascimento_et_al_2023], próteses veterinárias [F_Taub_2024], próteses faciais [F_Salazar_et_al_2022], aproximação facial forense [F_Sertalp_et_al_2024] e outros.
Durante todos esses anos, a ferramenta tem evoluído a partir da solução de problemas práticos e se convertido em uma estrutura confiável, dentre outras abordagens, para a reconstrução dos mais variados tipos de tomografias computadorizadas. Ao longo dos anos, o usuários têm encontrado problemas e enviado os DICOMs para depuração, isso permitiu ao sistema ter uma base de dados com indivíduos de todas as faixas etárias, distribuídos por 32 países (Estados Unidos da América, França, Espanha, Itália, Suíça, Dinamarca, Polônia, Alemanha, Peru, México, Argentina, Brasil, Chile, Colômbia, Malásia, Vietnã, Turquia, Índia, Marrocos, Tanzânia, Portugal, Moldávia, Honduras, Equador, Uruguai, Jordânia, Iraque, Israel, Uzbequistão, Tailândia, República Tcheca e Ucrânia).
Para mensurar a robustez da ferramenta de IA, foram testadas 151 tomografias computadorizadas, em uma amostra bem balanceada, com indivíduos de todas as faixas etárias, com as mais variadas deformações estruturais e defeitos faciais, tratando-se assim de um teste realmente rigoso para a solução tecnológica. Foram colhidos alguns dados relacionados ao item Manufacturer disponível nos DICOMs, resultando em 21 strings (“NewTom”, “PaloDEx Group”, “Gendex”, “ASAHIROENTGEN IND. CO., LTD.”, “J.Morita.Mfg.Corp.”, “GE MEDICAL SYSTEMS”, “Imaging Sciences International”, “SOREDEX”, “Xoran Technologies ®”, “Planmeca”, “HDXWILL”, “Carestream Health”, “Carestream Dental”, “MyRay”, “NIM”, “PreXion”, “Sirona”, “Dabi Atlante”, “Instrumentarium Dental” e “Vatech Company Limited”).
Os resultados foram surpreendentemente bons, das 151 tomografias testadas, todas foram reconstruídas, resultando em 0 erros neste quesito, o que pode ser muito útil ao usuário, uma vez que o DentalSegmentator efetua a reconstrução sem a necessidade do preenchimento dos fatores (thresahold). Além disso, ao final apresentou apenas dois erros de reconstrução dos dentes, mas um deles era relacionado a um conjunto de próteses e não a dentes efetivos. Já o único erro reportado em dentes efetivos, se deu em um indivíduo com dentes apenas na mandíbula, no entanto, o que chamou a atenção é que a ferramenta de IA não apresentou erro em situações muito mais críticas, como deformações estruturais ou dados faltantes, inclusive em tomografias parciais, indicando se tratar de uma abordagem significativamente robusta e confiável. Para resumir, em uma amostra de 151 tomografias heterogêneas, o nível de sucesso da ferramenta, dependendo da abordagem tomada, apresenta um índice na faixa de 98,68% - 99,34%.
Conclusão
Em face à intuitividade da ferramenta, bem como a sua robustez no amplo teste envolvendo 151 tomografias, a mesma está suficientemente desenvolvida para o uso no Linux e pronta para ser portada para o Windows e o MacOS.
Agradecimentos
Ao Dr. Davi Sandes Sobral, por ceder a tomografia computadorizada utilizada neste estudo. Um agradecimento especial a todos aqueles que enviaram tomografias computadorizadas, formando um grupo de arquivos importantíssimo para testar o uso do sistema em situações práticas.
Referências Bibliográficas
Cunha, H. S., da Costa Moraes, C. A., de Faria Valle Dornelles, R., & da Rosa, E. L. S. (2020). Accuracy of three-dimensional virtual simulation of the soft tissues of the face in OrtogOnBlender for correction of class II dentofacial deformities: an uncontrolled experimental case-series study. In Oral and Maxillofacial Surgery (Vol. 25, Issue 3, pp. 319–335). Springer Science and Business Media LLC. https://doi.org/10.1007/s10006-020-00920-0
Dot, G., Chaurasia, A., Dubois, G., Savoldelli, C., Haghighat, S., Azimian, S., Taramsari, A. R., Sivaramakrishnan, G., Issa, J., Dubey, A., Schouman, T., & Gajny, L. (2024). DentalSegmentator: Robust open source deep learning-based CT and CBCT image segmentation. In Journal of Dentistry (Vol. 147, p. 105130). Elsevier BV. https://doi.org/10.1016/j.jdent.2024.105130
Duarte, D. W., Fleith, I. J., Pinheiro, R. C., Machado, M. F., Zanin, E. M., & Collares, M. V. M. (2023). Mandibular morphology and distraction osteogenesis vectors in patients with Robin sequence. In International Journal of Oral and Maxillofacial Surgery (Vol. 52, Issue 4, pp. 442–450). Elsevier BV. https://doi.org/10.1016/j.ijom.2022.07.007
Façanha de Carvalho, E., Alkmin Paiva, G. L., Yonezaki, F., & Machado, G. G. (2021). Computer-Aided Surgical Simulation in Severe Atrophic Mandibular Fractures: A New Method for Guided Reduction and Temporary Stabilization Before Fixation. In Journal of Oral and Maxillofacial Surgery (Vol. 79, Issue 4, p. 892.e1-892.e7). Elsevier BV. https://doi.org/10.1016/j.joms.2020.11.011
Isensee, F., Jaeger, P. F., Kohl, S. A. A., Petersen, J., & Maier-Hein, K. H. (2020). nnU-Net: a self-configuring method for deep learning-based biomedical image segmentation. In Nature Methods (Vol. 18, Issue 2, pp. 203–211). Springer Science and Business Media LLC. https://doi.org/10.1038/s41592-020-01008-z
Lobo, F., Filho, L. I., Sigua-Rodriguez, E. A., da Silva, B. G., Tolentino, E. de S., Borges, Y. M., da Silva, M. C., Tonin, R. H., & Iwaki, L. C. V. (2022). Evaluation of ortogonblender software bone movement tools in bimaxillary orthognatic surgeries performed in dolphin software. In Journal of Stomatology, Oral and Maxillofacial Surgery (Vol. 123, Issue 4, pp. 417–421). Elsevier BV. https://doi.org/10.1016/j.jormas.2021.10.001
Moraes, C., & Dakir, I. (2021). Segmentação Automática dos Dentes com o MeshSegNet e o OrtogOnBlender. figshare. https://doi.org/10.6084/M9.FIGSHARE.14850105. https://ortogonline.com/doc/pt_br/OrtogOnLineMag/3/SegmentaDentes.html
Moraes, C., da Rosa, E., & Dornelles, R. (2020). Ortogonblender - O Que É e aspectos técnicos. Retrieved from https://www.ciceromoraes.com.br/doc/pt_br/OrtogOnBlender/OrtogOnBlender_Como_Funciona.html
Moraes, C., Schreiner, T., Startek, B., & Beaini, T. L. (2024). Alinhamento em Frankfurt, Colocação de Pontos Anatômicos e Segmentação do Crânio por Inteligência Artificial no OrtogOnBlender. figshare. https://doi.org/10.6084/M9.FIGSHARE.25140020. https://ortogonline.com/doc/pt_br/OrtogOnLineMag/8/AI.html
Moraes, C., Dakir, I., Startek, B., Dornelles, R., & Rosa, E. da. (2024). Ferramenta de Importação de Tomografia Computadorizada no OrtogOnBlender XP. figshare. https://doi.org/10.6084/M9.FIGSHARE.27648876. https://ortogonline.com/doc/pt_br/OrtogOnLineMag/10/CT_Scan.html
Nascimento, B. C. G., Moraes, C. A. D. C., Neto, R. P., Rocha, B. A., Miranda, E. D. P., Bessa, J. D., Nahas, W. C., Hallak, J., Mulhall, J. P., & Gomes, C. M. (2023). (167) 3D Penile Reconstruction Imaging in Complex Peyronie’s Disease (PD): Proof of Concept Study. In The Journal of Sexual Medicine (Vol. 20, Issue Supplement_1). Oxford University Press (OUP). https://doi.org/10.1093/jsxmed/qdad060.162
Salazar-Gamarra, R., Cárdenas-Bocanegra, A., Masch, U., Da Costa Moraes, C. A., Seelaus, R., Lopes Da Silva, J. V., & Lauria Dib, L. (2022). Color translation from monoscopic photogrammetry +ID Methodology into a Polyjet final 3D printed facial prosthesis. In F1000Research (Vol. 11, p. 582). F1000 Research Ltd. https://doi.org/10.12688/f1000research.111196.1
Sertalp, E., Moraes, C., & Bütün, E. (2024). Facial reconstruction of a deformed skull from the Roman period of Juliopolis. In Heritage Science (Vol. 12, Issue 1). Springer Science and Business Media LLC. https://doi.org/10.1186/s40494-023-01124-x
Sobral, D. S., Duarte, D. W., Dornelles, R. F. V., & Moraes, C. A. C. (2021). 3D Virtual Planning for Rhinoplasty Using a Free Add-On for Open-Source Software. In Aesthetic Surgery Journal (Vol. 41, Issue 8, pp. NP1024–NP1032). Oxford University Press (OUP). https://doi.org/10.1093/asj/sjab085
Taub, B. (2024). Injured tortoise zooms around on new wheels thanks to 3D-printed prosthesis. IFLScience. https://bit.ly/4eWxmKN