Alinhamento em Frankfurt, Colocação de Pontos Anatômicos e Segmentação do Crânio por Inteligência Artificial no OrtogOnBlender¶
Atenção
Este material utiliza a seguinte licença Creative Commons: Atribuição 4.0 Internacional (CC BY 4.0).
Introdução¶
A inteligência artificial (IA) passou a chamar muita atenção na imprensa mainstream a partir do ano de 2019, quando a popularização da tecnologia se fez mais evidente. Outrossim, há muito as suas bases vêm sendo estabelecidas, uma vez que já em 1843, por exemplo, Lady Ada Lovelace, a criadora da primeira linguagem de computador não tinha certeza sobre a possibilidade das máquinas não pensarem por si só, indicando que apenas seria possível sabê-lo, depois que construíssem e testassem tais máquinas [B_McCorduck_1977]. Infelizmente a Máquina Analítica de Babbage acabou não sendo construída, mas os esforços relacionados a computação e a IA, ainda que de modo indireto, permaneceram latentes. Mais de cem anos depois, Alan Turing desenvolve o Teste de Turing, que seria a base para a avaliação da eficácia da IA em relação a comunicação humana. Na mesma década, em 1956 John McCurthy oficializa o termo Inteligência Artificial, definindo-o como “a ciência e a engenharia de criação de máquinas inteligentes”. Na década posterior, em 1964 Eliza o primeiro chatbot é criado por Joseph Weizenbaum e em 1966, Shakey, o primeiro robô móvel com capacidade de perceber e raciocinar sobre o que o rodeia é apresentado ao mundo. Nem tudo correu bem na história da IA, uma vez que dois grandes invernos nortearam o seu desenvolvimento, o primeiro no final da década de 1970, por conta das limitações técnicas e o segundo no final dos anos 1980, desta vez, devido ao custo excessivo das implementações [B_Kaul_2020]. Os anos 1990 trariam a tecnologia novamente às notícias, graças as históricas partidas entre o supercomputador da IBM, Deep Blue versus o então campeão mundial de xadrez, o russo Garry Kasparov, onde o segundo foi derrotado em 1997, marcando uma virada histórica, posto que no ano anterior o russo tinha levado a melhor. Depois de perder, este saiu da sala rapidamente, explicando o motivo posteriormente, quando disse: “Eu sou humano, quando vejo algo que está muito além da minha compreensão, tenho medo” [B_Ekmekci_2020]. Na esteira do sucesso no desenvolvimento, a mesma IBM lança em 2007 um sistema de respostas a perguntas, o Watson [B_Kaul_2020], que em 2011 fez história ao derrotar os dois maiores campeões de todos os tempo do programa televisivo Jeoparty! [B_IBM_2011]. Com a evolução do processo de linguagem natural, os chatbots de comunicação superficial foram se desenvolvendo e converteram-se em interfaces com grande poder de comunicação e sucesso comercial, como a Siri da Apple (2011) e a Alexa da Amazon (2014) [B_Kaul_2020]. Já no final da década de 2010 e início de da de 2020, houve uma significativa popularização de ferramentas baseadas em IA, como o Chat GPT (https://chat.openai.com/), o Bard da Google (https://bard.google.com/chat) e outras.
O presente desenvolvimento da IA inspirou uma série de especialistas a focarem os seus esforços para o campo da saúde humana, especificamente nas áreas da radiologia, odontologia, medicina e afins. Em 2021 o OrtogOnBlender (OOB), um add-on para o planejamento de cirurgias faciais (https://www.ciceromoraes.com.br/doc/pt_br/OrtogOnBlender/index.html), adicionou em seu sistema um processo de segmentação dentária baseada em inteligência artificial [B_Moraes_2021], que apesar de funcionar bem em modelos limpos, se mostrou problemático na segmentação de arcadas com braquetes, presentes na maioria dos casos de planejamento cirúrgico. Seguindo os estudos relacionados à IA, os desenvolvedores do OOB buscaram outras alternativas, não apenas para a segmentação dos dentes, mas dos ossos faciais, alinhamento automático do crânio e reconhecimento de pontos anatômicos. Uma ferramenta que se mostrou potencialmente útil para tais tarefas é a SlicerAutomatedDentalTools (https://github.com/DCBIA-OrthoLab/SlicerAutomatedDentalTools), desenvolvida como um módulo para o software Slicer (https://www.slicer.org/), mas que permite uma comunicação bastante eficaz com o Blender, via Python script. Por ser de código aberto e rodar em sistemas operacionais diferentes, tornou-se uma opção perfeita para ser inserida no OOB e ser testada como uma ferramenta que tornará o processo de planejamento mais rápido, sem abrir mão da acurácia necessária para tal.
O objetivo deste trabalho é relatar como as ferramentas de inteligência artificial para o reposicionamento do volume alinhado ao plano de Frankfurt, a demarcação de pontos craniométricos e a segmentação de estruturas faciais de maneira automática, foram adicionadas à interface do add-on OrtogOnBlender.
Materiais e Métodos¶
O SlicerAutomatedDentalTools é composto por uma série de ferramentas/bibliotecas:
AMASSS (Automatic Multi-Anatomical Skull Structure Segmentation), uma ferramenta para segmentação óssea que treinou o seu modelo utilizando 618 tomografias CBCT, resultando em uma pontuação Dice 0.962±0.02. É importante pontuar que dentre as tomografias utilizadas para o treinamento, estavam presentes as de pacientes com fissura labial e/ou fenda palatina, o que aumentou a complexidade da predição e ampliou as possibilidades de uso da ferramenta [B_Gillot_2022].
ALI-CBCT (Automated Landmark Identification for Cone-Beam Computed Tomography), uma ferramenta para a localização de pontos anatômicos que utilizou com base de trinamento 143 tomografias CBCT de centros radiológicos e países diferentes. No total foram utilizados 119 pontos distribuídos pela superfície do crânio, resultando em uma média de erro de 1.54±0.87 mm [B_Gillot_2023].
ASO-CBCT (Automated Standardized Orientation for Cone-Beam Computed Tomography), uma ferramenta para alinhamento do crânio, treinada com 39 modelos, cuja média de erro foi 0.089 quando 0 é o alvo [B_Anchling_2020].
O trabalho de programação consistiu em portar todos os passos efetuados manualmente no módulo SlicerAutomatedDentalTools do Slicer, para um script escrito em Python, para que o acionamento do processo fosse feito no Blender, via OrtogOnBlender. O objetivo primordial foi facilitar a experiência do usuário e reduzir um longo processo em apenas dois passos: 1) Selecionar o diretório da tomografia computadorizada e 2) Computar os dados. Deste modo, foi necessário fazer uma série de edições nos scripts baixados e instalados a partir do Gerenciador de Módulos do Slicer. A solução foi testada em três sistema operacionais diferentes, sendo o Linux, o Windows e o MacOS, visando contemplar todos os usuários do OOB.
O trabalho com o script consistiu nas seguintes etapas:
Conversão do arquivo DICOM para uma extensão compatível (arquivo único *.nii.zg);
Alinhamento da tomografia no plano horizontal de Frankfurt;
Posicionamento dos 119 pontos anatômicos e importação dos mesmos na cena;
Segmentação, importação e aplicação de shader (material) na mandíbula, maxila, via respiratória, cervical e parte da região superior do crânio;
Geração de uma sequência DICOM com o volume alinhado ao plano de Frankfurt, bem como a sua importação na cena e configuração dos materiais.
Foram testadas 30 tomografias aleatórias Fan-beam e Cone-beam, para que o tempo médio do processo fosse estabelecido. A mensuração temporal foi efetuada em uma máquina com as seguintes características:
Resultados e Discussão¶
Depois de uma série de testes efetuados em três sistemas operacionais diferentes (Linux, Windows e MacOS), foi gerada uma documentação unificada, para que os interessados pudessem baixar os programas e configurar o ambiente objetivando permitir o pleno funcionamento do SlicerAutomatedDentalTools no OOB. Tal documento pode ser encontrado no seguinte link: https://docs.google.com/document/d/1th6xnD9Xg7qo9-Guxpdu_3j3jV_t-E92XWBRkMnKbzo/edit?usp=sharing.
Por conta do grande volume dos arquivos e, pelo teste inicial ter sido efetuado em poucos computadores, a documentação da instalação está dividida em muitos passos, de modo que fique mais fácil depurar eventuais problemas encontrados pelos usuários para finalizar o processo.
Atenção
O funcionamento dos comandos do script depende da instalação acima, caso contrário não será possível seguir com os testes, ainda que os botões se façam visíveis na interface do OrtogOnBlender.
Fig. 14 Interface do SlicerAutomatedDentalTools no OthersOnBlender.¶
A proposta inicial da interface gráfica é ser o mais simples possível, limitando-se a dois elementos, um seletor de diretório para indicar onde se encontra a tomografia computadorizada e um botão para ordenar todos os processos envolvidos (Fig. 14).
O add-on apresenta a informação do tempo necessário para todo o processo e testes foram efetuados em 50 tomografias, tanto Fan-beam quanto Cone-Beam. Em linhas gerais o tempo médio para o computo de dados em uma Fan-beam é de 24 minutos, já em uma Cone Beam a média caiu para 19 minutos.

Fig. 15 Resultado do processamento: Tecido mole em evidência.¶
Após o processamento, avista-se a face (tecido mole) envolto pelo que parece ser uma caixa, resultado do alinhamento ao plano horizontal de Frankfurt (Fig. 14).
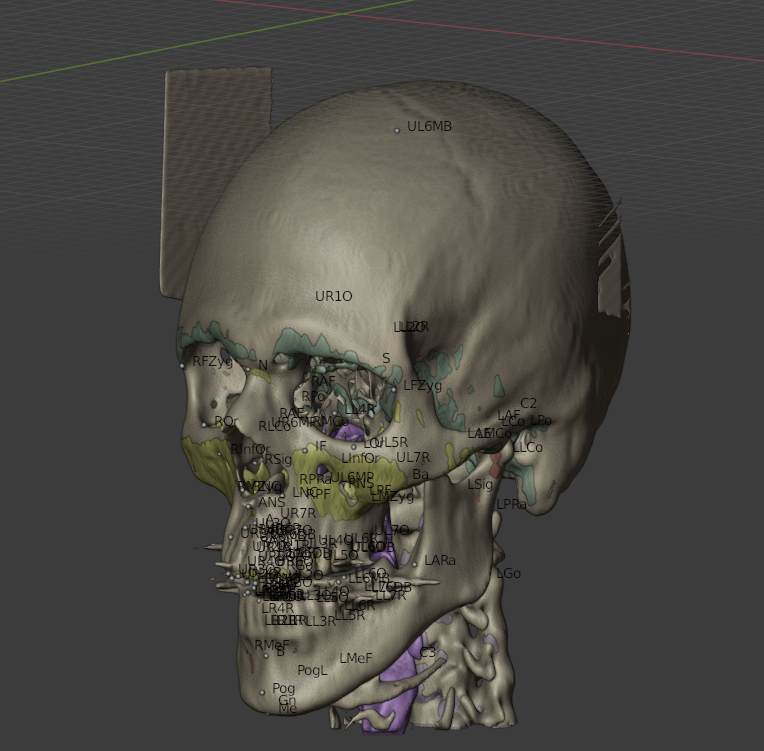
Fig. 16 Resultado do processamento: Ossos em evidência.¶
Ao se ocultar o tecido mole, é possível ver o crânio completo, tanto a face quanto o crânio (e os dentes) foram importados utilizando o VTK-ITK [B_Moraes_2021b] do OOB, o entanto a tomografia alvo não foi a original, mas aquela alinhada ao plano de Frankfurt pela ferramenta ASO. O objetivo de importar a reconstrução efetuada pela ferramenta “nativa” do OOB é fornecer um elemento de comparação e potencial validação para a malha advinda da segmentação efetuada pelo AMASSS. Nos testes efetuados, em relação aos ossos, ambas se compatibilizaram visualmente (Fig. 16), mas um estudo mais aprofundado, focando tal abordagem se fará necessário.
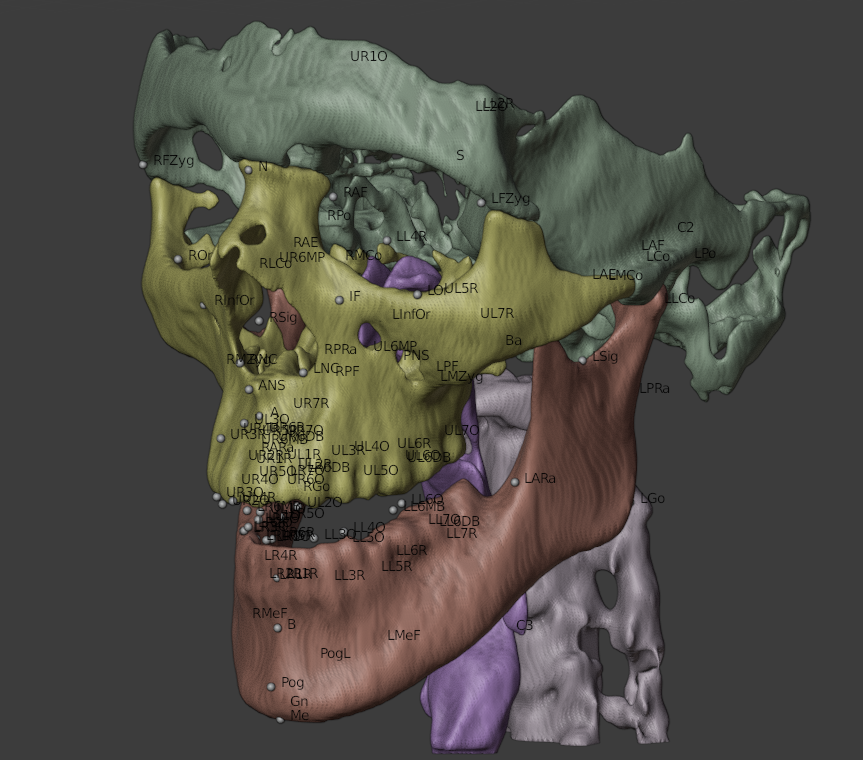
Fig. 17 Resultado do processamento: Ossos segmentados e pontos posicionados.¶
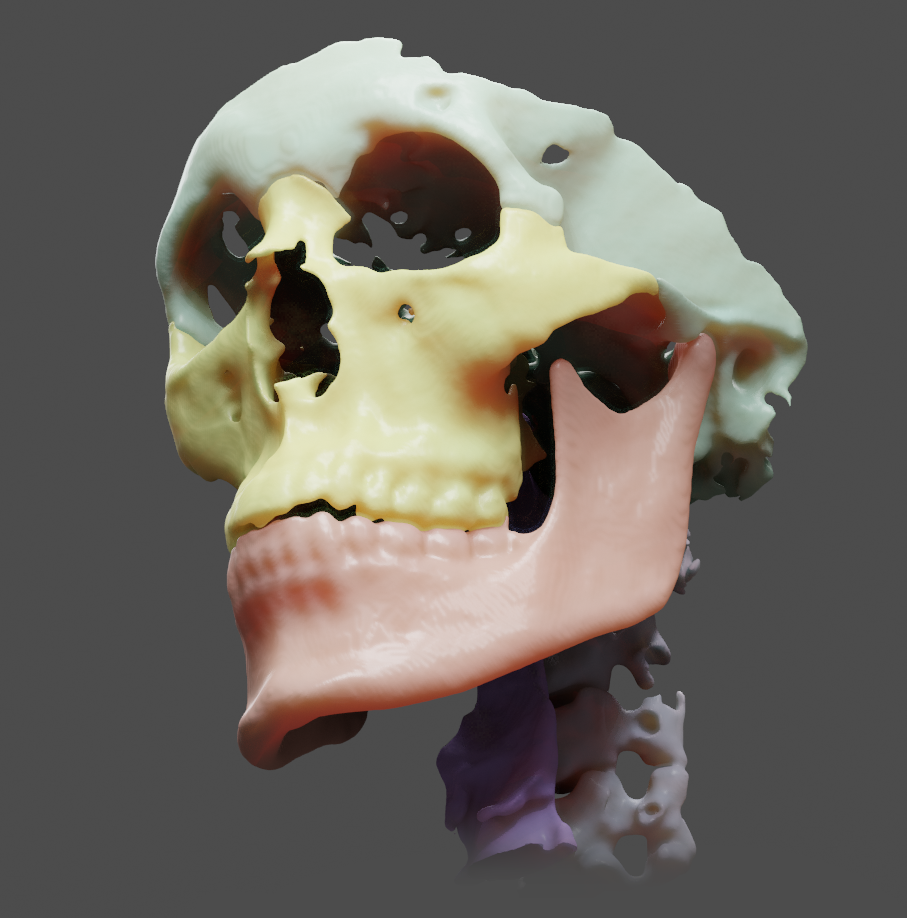
Os ossos segmentados receberam as mesmas cores da importação do Slicer, com exceção da via respiratória (Fig. 17). Parece haver uma pequena perda de dados nos dentes, o que poderia acarretar em problemas de alinhamento com o modelo digitalizado em 3D, mas a importação advinda do VTK-ITK é o suficiente para resolvê-lo. A mandíbula também parece um pouco mais fina, mas isso também não seria um problema, pois mesmo que não refletisse o a espessura real (acurácia de 100%), poderia ser utilizada como parâmetro de booleana para a segmentação da peça, desde que recebesse um pequeno offset para exceder os limites do modelo recontruído pelo VTK-ITK.

Fig. 18 Resultado do processamento: Pontos em evidência.¶
Os pontos foram importados como esferas de 2mm de diâmetro (Fig. 18). Embora a ferramenta apresentasse uma performance muito boa, em todos os testes efetuados, ocorriam falhas em relação a alguns pontos anatômicos que, ou não eram encontrados, ou quando encontrados poderiam estar em uma posição equivocada. Usuários do OOB se manifestaram e indicaram que poucos dos pontos são realmente utilizados nos trabalhos diários, de modo que, ficou acordada uma futura organização acerca do grupo, ou grupos de pontos que serão selecionados no momento do cálculo. Outra preocupação manifestada foi a precisão dos pontos, ainda que os autores indiquem uma baixa margem de erro, testes precisam ser efetuados durante o processo de planejamento cirúrgico para atestar a precisão tanto do posicionamento de pontos, como o do alinhamento e da segmentação.
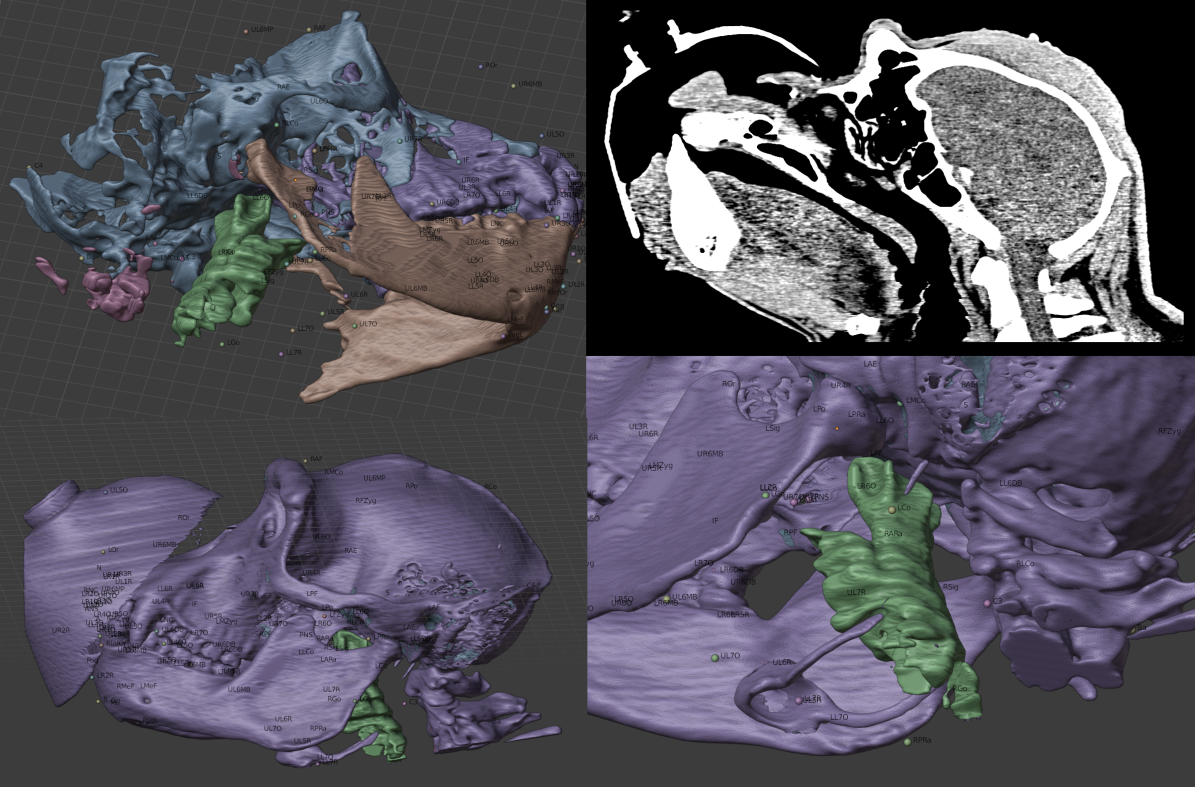
Fig. 19 Tentativa de processamento na tomografia de um Pan troglodytes, reusltando na segmentação parcial da mandíbula e total da via respiratória.¶
No entanto, mesmo quando a precisão dos pontos não é tão grande, ao menos indicam uma região relacionada ao crânio, podendo servir como elemento delimitador em trabalhos de levantamento de dados pré e pós cirúrgicos, dentre outros. As ferramentas de pontos e segmentação funcionaram muito bem com adultos, de modo significativamente bom com crianças e regionalmente satisfatórios em bebês. Até em testes de segmentação com tomografias de gorilas e chimpanzés a ferramenta de segmentação permitiu a separação parcial da mandíbula e total da via respiratória (Fig. 19), ainda que o objetivo da ferramenta não fosse reconstruir outros primatas.

Fig. 20 Iluminação da cena.¶
A iluminação e renderização em tempo real da cena (Fig. 20) foi efetuada com a ferramenta ILLUMINATE!, disponível na aba ForensicOnBlender, seção Soft Tissue Markers.
Conclusão¶
A programação via Python scrip permitiu trabalhar com as ferramentas AMASSS, ALI-CBCT e ASO-CBCT por chamada direta da interface do Blender 3D, utilizando o ambiente do OrtogOnBlender. Caso se mostrem precisos, o alinhamento ao plano de Frankfurt, a segmentação da mandíbula e o posicionamento de pontos, efetuados de modo automático, vão reduzir significativamente o tempo dos planejamento cirúrgicos, trazendo mais agilidade para essas tarefas digitais. Os autores esperam que a presente publicação instigue os usuários a instalarem, testarem e definirem parâmetros para o uso das ferramentas aqui demonstradas, que sem dúvida, são muito promissoras e parecem representar o futuro do planejamento cirúrgico.
Agradecimentos¶
Ao Dr. Richard Gravalos por ceder a tomografia utilizada para ilustrar o presente estudo e claro, aos desenvolvedores das ferramentas abordadas, por disponibilizarem-as de modo gratuito e com o código aberto.
Referências Bibliográficas¶
Ekmekci, P. E., & Arda, B. (2020). History of Artificial Intelligence. In Artificial Intelligence and Bioethics (pp. 1–15). Springer International Publishing. https://doi.org/10.1007/978-3-030-52448-7_1
Gillot, M., Baquero, B., Le, C., Deleat-Besson, R., Bianchi, J., Ruellas, A., Gurgel, M., Yatabe, M., Al Turkestani, N., Najarian, K., Soroushmehr, R., Pieper, S., Kikinis, R., Paniagua, B., Gryak, J., Ioshida, M., Massaro, C., Gomes, L., Oh, H., … Prieto, J. C. (2022). Automatic multi-anatomical skull structure segmentation of cone-beam computed tomography scans using 3D UNETR. In S. V E (Ed.), PLOS ONE (Vol. 17, Issue 10, p. e0275033). Public Library of Science (PLoS). https://doi.org/10.1371/journal.pone.0275033
Gillot, M., Miranda, F., Baquero, B., Ruellas, A., Gurgel, M., Al Turkestani, N., Anchling, L., Hutin, N., Biggs, E., Yatabe, M., Paniagua, B., Fillion‐Robin, J., Allemang, D., Bianchi, J., Cevidanes, L., & Prieto, J. C. (2023). Automatic landmark identification in cone‐beam computed tomography. In Orthodontics & Craniofacial Research (Vol. 26, Issue 4, pp. 560–567). Wiley. https://doi.org/10.1111/ocr.12642
IBM. (2011). Watson, jeopardy! champion. https://www.ibm.com/history/watson-jeopardy
Kaul, V., Enslin, S., & Gross, S. A. (2020). History of artificial intelligence in medicine. In Gastrointestinal Endoscopy (Vol. 92, Issue 4, pp. 807–812). Elsevier BV. https://doi.org/10.1016/j.gie.2020.06.040
Anchling, L. (2020). Mic Project Weeks. NA-MIC. https://projectweek.na-mic.org/PW38_2023_GranCanaria/Projects/ASO_CBCT/
McCorduck, P., Minsky, M., Selfridge, O.G., & Simon, H.A. (1977). History of Artificial Intelligence. International Joint Conference on Artificial Intelligence.
Moraes, C., & Dakir, I. (2021). Segmentação Automática dos Dentes com o MeshSegNet e o OrtogOnBlender. figshare. https://doi.org/10.6084/M9.FIGSHARE.14850105. https://ortogonline.com/doc/pt_br/OrtogOnLineMag/3/SegmentaDentes.html
Moraes, C., Dakir, I., Dornelles, R., & Da Rosa, E. (2021). Reconstrução de Tomografias com o VTK Python, o SimpleITK e o Multiprocessing. figshare. https://doi.org/10.6084/M9.FIGSHARE.14370902. https://ortogonline.com/doc/pt_br/OrtogOnLineMag/3/VTK_ITK_multi.html