Conversão de Imagens em Arquivos DICOM com o OrtogOnBlender¶
ISBN: 978-65-00-29233-6
O presente capítulo tem por objetivo apresentar a conversão de imagens .JPG em arquivos DICOM utilizando interface gráfica disponível no OrtogOnBlender.
Atenção
Este material utiliza a seguinte licença Creative Commons: Atribuição 4.0 Internacional (CC BY 4.0).
Apresentação¶
As tomografias computadorizas permitem ao usuário acessar digitalmente as regiões internas de um corpo ou objeto. Há uma grande oferta de serviços principalmente voltados a clínicas de tratamento humano, mas a cada dia aumenta o número de campos que se beneficiam da tomografia computadorizada e suas novas tecnologias, como por exemplo a arqueologia e a biologia. No caso da biologia, as micro tomografias ou micro CT-Scan, permitem a digitalização de pequenos animais e a observação detalhada de suas anatomias. Não são todos os centros de pesquisa que possuem o acesso a esses equipamentos, mas graças à internet e a portais como o YouTube ou o Digimorph, é possível analisar materiais gerados por terceiros. Os vídeos disponibilizados de modo online podem ser baixados e convertidos em imagens e estas por sua vez, em arquivos DICOM, o que pode evoluir para uma reconstrução 3D da estrutura.
Há também um número significativo de exames tomográficos que são disponibilizados como imagens .JPG ou .TIF e ainda aqueles mais antigos cuja a única fonte é uma impressão em folha ou filme. Em ambos os casos o material pode ser escaneado, segmentado (recortado) e convertido em arquivo DICOM.
Aviso
Este capítulo é direcionado a usuários contumazes do OrtogOnBlender, de modo que não serão explanados conceitos básicos relacionados ao add-on, focando principalmente nos resultados do desempenho.
Como Funciona¶
As ferramentas de conversão de vídeo em imagens e de imagens em DICOM já estão disponíveis no OrtogOnBlender há um bom tempo, no entanto, a solução apresentava uma série de bugs e não funcionava no sistema operacional Mac OS X, apenas no Linux e no Windows.
O problema foi resolvido ao se utilizar para o conversão o aplicativo img2dcm, as biblioteca Pydicom e Python SimpleITK e também substuindo o VolView, já defasado, pelo Slicer, uma solução efetivamente multiplataforma.
Atenção
A subistituição do VolView se deveu exclusivamente ao fato do mesmo não oferecer instalação para sistemas Mac OS X mais modernos, comprometendo o uso multiplataforma do OrtogOnBlender. A ferramenta é robusta e pode ser muito útil na conversão de uma série de arquivos de tomografias digital, principalmente aqueles pertencentes a máquinas mais antigas. O programa ainda se faz presente na instalação do OrtogOnBlender para o Linux e o Windows, podendo ser acessado no diretório de principal.
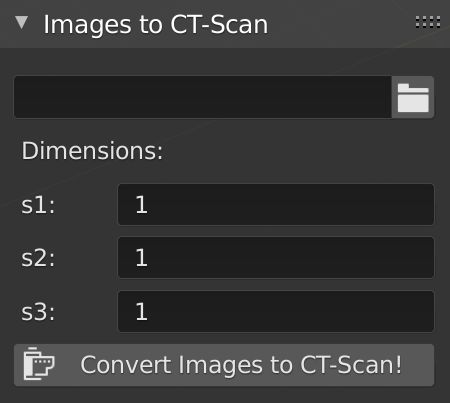
Dimensões do voxel.¶
A ferramenta se encontra na seção Images to CT-Scan na aba Others posicionada à direita na 3D View. Ao expandir a seção a primeira parte é composta pelo campo onde o usuário indicará o diretório com a sequência de imagens (Fig. 1). Em Dimensions é informado o valor de cada pixel, por padrão é 1mm, ou seja, se for reconstruída um voxel de 100x100x100 o tamanho da matriz será de 10cmx10cmx10cm.
Ao clicar em Convert Images to CT-Scan o arquivo de tomografia é gerado e aberto pelo Slicer.
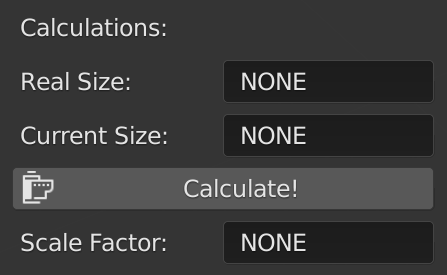
Cálculo do valor do pixel.¶
Quase sempre é necessário adequar o valor dos eixos para colocar a tomografia na escala (Fig. 2). Essa parte da ferramenta permite ao usuário informar o valor real do objeto (Real Size), o valor atual da medida (Current Size), e através de um cálculo (Calculate!) receber o valor do pixel no eixo (Scale Factor).
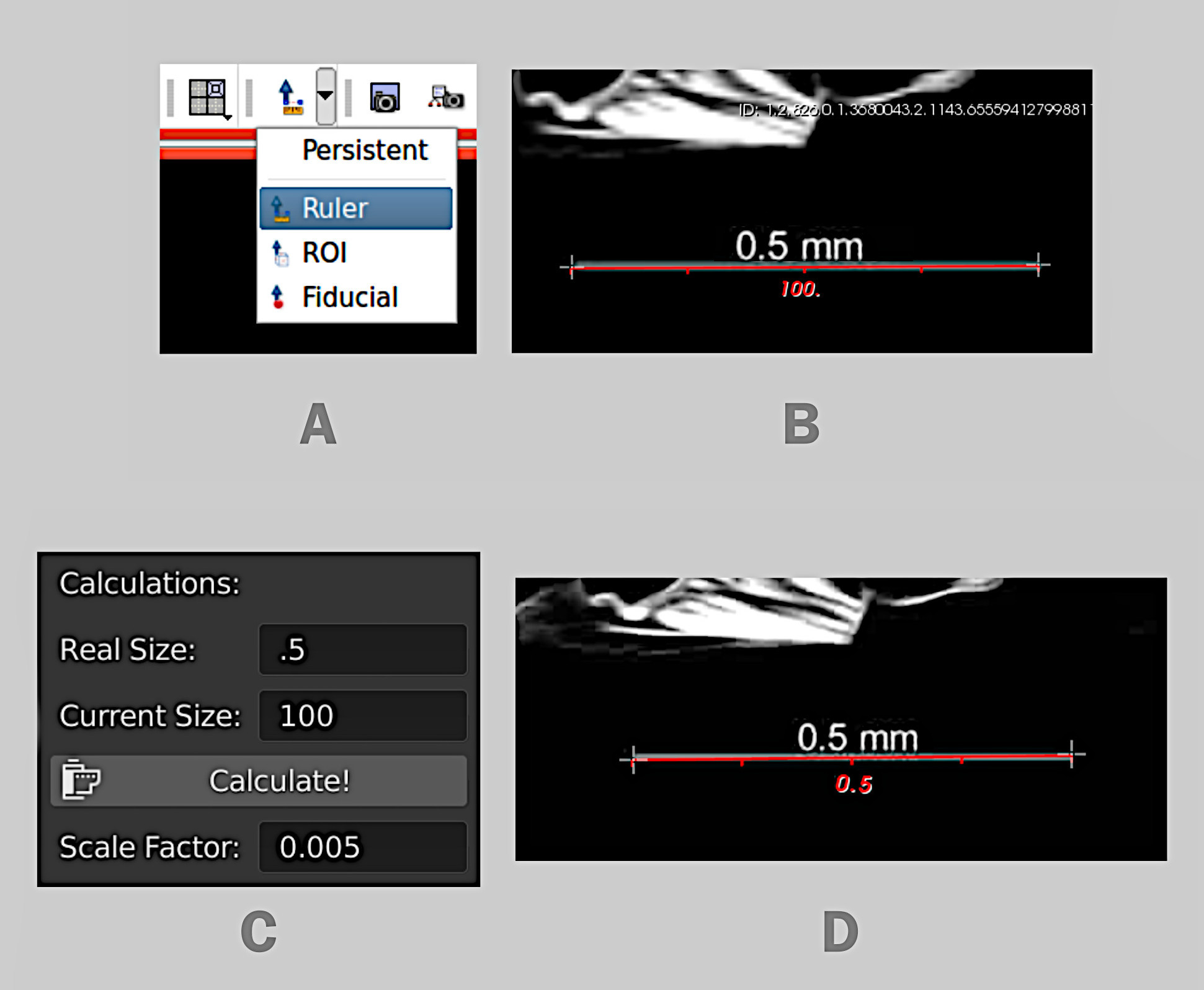
Passo-a-passo do cálculo do valor do pixel.¶
Como exemplo, o usuário gera a tomografia que é aberta no Slicer. Naquele software há a ferramenta Ruler (régua) na parte superior da interface (Fig. 3, A). Assim que a ferramenta é acionada, basta clicar em dois pontos de uma medida conhecida. O exemplo conta com uma escala apresentando 0.5 mm, que ao ser medida revela-se na verdade 100 mm (Fig. 3, B). No OrtogOnBlender o cálculo do fator é feito, colocando o Real Size, ou seja, o tamanho real que é 0.5, em seguida o Current Size, que é o tamanho atual, ou seja 100 mm. Ao clicar em Calculate! o valor resultante é 0.005 (Fig. 3, C). O usuário então fechará o Slicer, voltará ao OrtogOnBlender, corrigirá esse valor na parte superior da interface (ver Fig. 1), rodará novamente o gerador de DICOM (Convert Images to CT-Scan) e ao fazer novamente a medida, se tudo der certo o valor resultante será o mesmo da escala (Fig. 3, D).
Dica
Há um material amplamente explanativo disponível na documentação oficial do OrtogOnBlender, trata-se do capítulo intitulado Convertendo um Vídeo em um Arquivo DICOM [A20]. Lá é descrito todo o processo de conversão, desde o download de um vídeo no YouTube, passando pela conversão em uma sequência de imagens, a conversão da imagens em arquivos DICOM e finalmente a reconstrução 3D da captura.
Aviso
Recomanda-se fortemente utilizar a ferramenta VTK + ITK [A18] para a reconstrução 3D dos arquivos DICOM gerados a partir de imagens.
Estudo de Caso¶
Uma pequena peça óssea foi digitalizada e disponibilizada como uma série de 1696 arquivos .JPG de 1528x1528 pixels. A informação prévia é de que se tratava de uma tomografia de proporção 1x1x1, ou seja, o voxel apresenta o mesmo fator em todos os eixos.
Aviso
Os nomes dos arquivos de imagens não podem conter espaços sob pena de gerar erro no momento da conversão. Em face disto, recomenda-se ao usuário renomear a sequência, caso seja necessário, evitando além dos espaços, caracteres especiais como o ç, ã, í, etc.

Medida dos pontos mais extremos da peça.¶
Foi efetuada uma medida nas extremidades mais distantes e sólidas da peça, de modo a extrair um valor conhecido. O resultado foi 5.33 mm (Fig. 4).
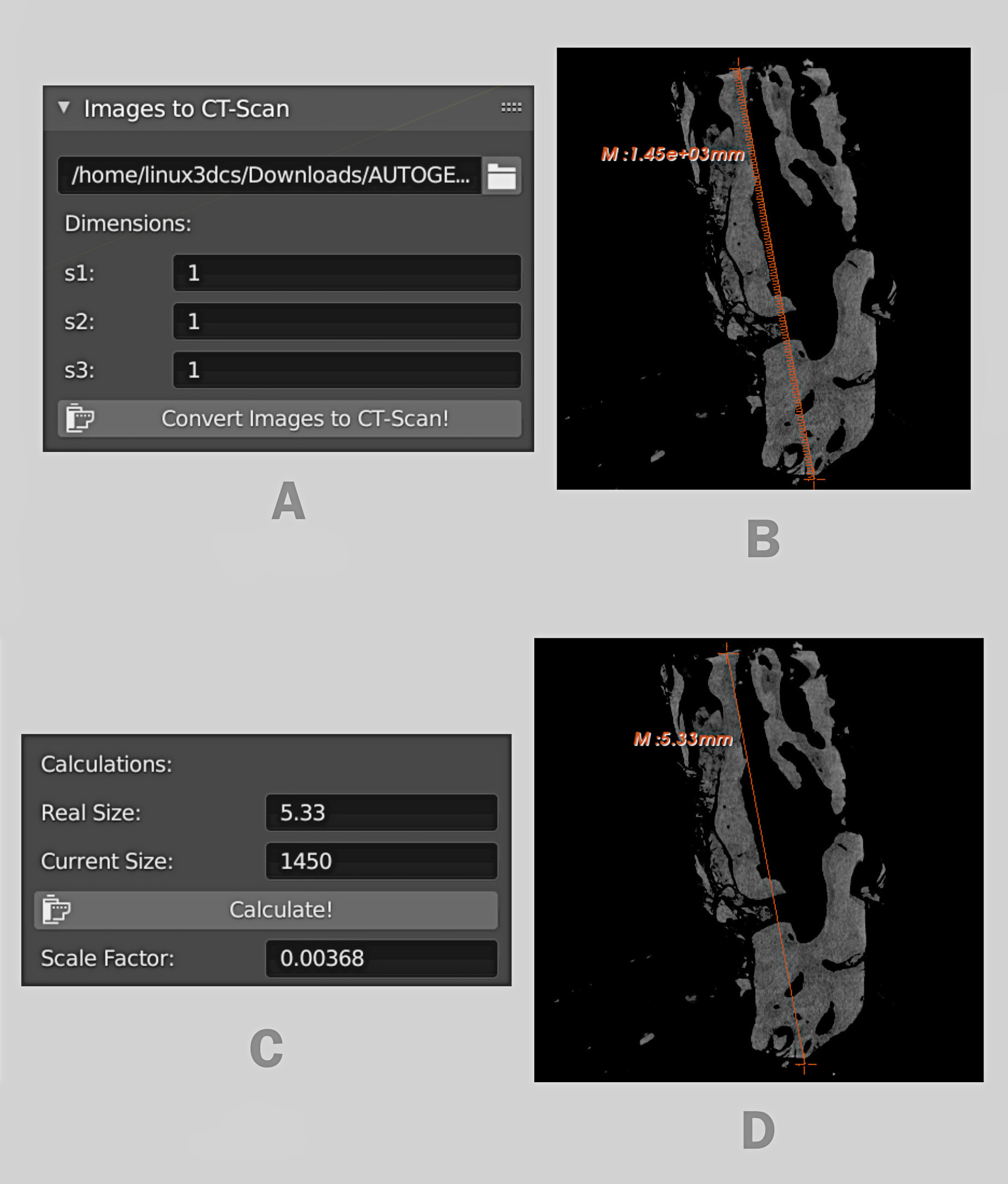
Correção da escala.¶
Gerou-se uma tomografia mantendo os valores padronizados da ferramenta (Fig. 5, A). Assim que a tomografia foi aberta no Slicer, com a ferramenta régua, mensurou-se as extremidades da peça, resultando no valor 1.45e-03mm (Fig. 5, B), que corresponde a 1.45^10³, ou seja, 1450mm. Para corrigir a distância, que corresponde a 1,45m, recorreu-se a parte inferior da ferramenta, onde foram indicadas a distância real (5.33) e a distância atual (1450), resultando no fator 0.00368 após o cálculo (Fig. 5, C). Gerada novamente a tomografia e procedida a medida, atestou-se que o voxel estava na escala (Fig.5, D).
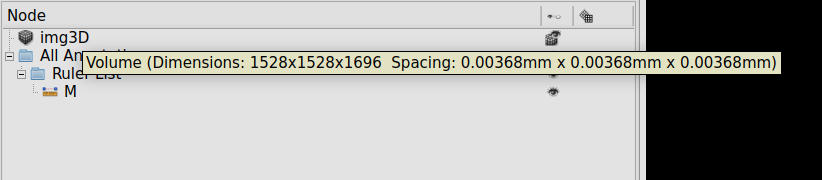
Dados da matriz 3D.¶
Ainda no Slicer e recorrendo ao módulo Data, é possível acessar os dados da tomografia e nesse caso, tem-se uma matriz de 1528 x 1528 x 1696, ou seja, trata-se de 1696 imagens sobrepostas, cada uma com as dimensões de 1528x1538px. Em seguida aparecem três outros valores, 0.00368mm x 0.00368mm x 0.00368mm (Fig. 6). Isso significa que o voxel, grosso modo, o “pixel 3D” da imagem tem essas três dimensões. Então, se temos 1528px de um lado, vezes o fator do voxel naquele lado, ou seja 0,00368, resultará em 5,62 mm. É possível aferir que cada imagem tem 5,62 x 5,62 mm, mas ainda falta o altura da tomografia, composta pelas 1696 fatias. Se já conta-se com o valor do espaço, basta multiplicar, nesse caso: 1696 x 0,00368 = 6,24 mm. Logo, temos uma tomografia de 5,62 x 5,62 x 6,24 mm.
As dimensões da tomografia são muito pequenas, mas a resolução da mesma é muito grande, se multiplicados todos os lados (1.528x1.528x1.696 = 3.959.793.664) o resultado será quase 4 trilhões de voxels!
Por padrão o OrtogOnBlender trabalha com matrizes limitadas em 512 pixels, multiplicando todos os lados (512x512x512 = 134.217.728) chaga-se ao valor de pouco mais de 134 milhões de voxels, ou seja, 3,39% do volume da tomo aqui abordada. Isso certamente vai permitir que o computador processe a informação de modo muito mais rápido. A questão é como reduzir a resolução?
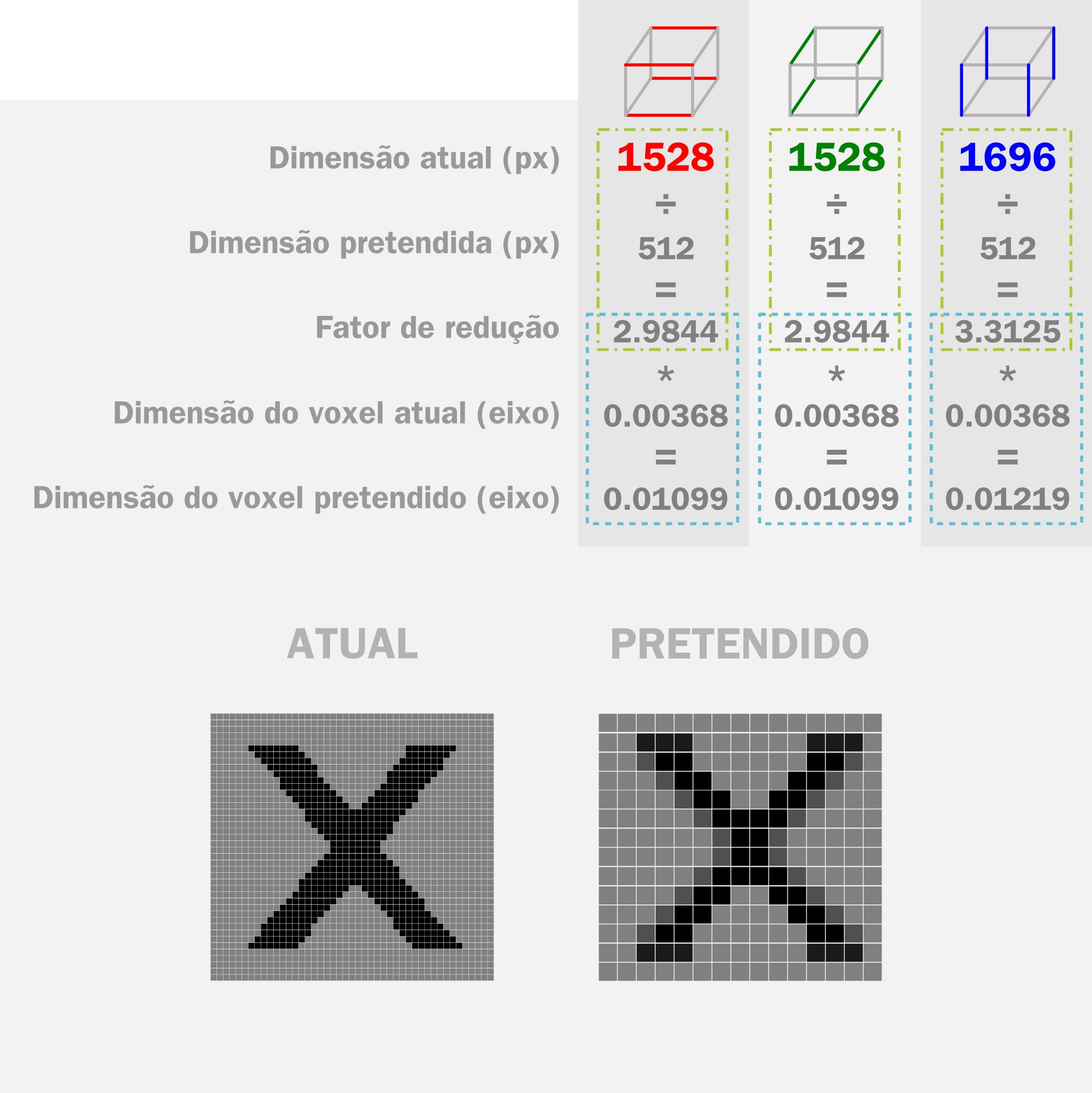
Cálculo de redução da resolução.¶
O cálculo para reduzir a resolução é simples, basta tomar o valor atual dos pixels em um eixo, dividir pelos pixels pretendidos e depois multiplicar o resultado pela dimensão atual do eixo do voxel. Se tomarmos, por exemplo, o eixo com 1528 px teremos o seguinte resultado para o fator de redução: 1528 ÷ 512 = 2,9844. O valor de 2,9844 trata-se do fator que será utilizado para aumentar o tamanho do voxel em um eixo, se atualmente o valor dele é 0,00368 basta multiplicá-lo pelo fator em questão: 2,9844 * 0,00368 = 0,01099.
Pode parece confuso em um primeiro momento para aqueles que não estão acostumados com o uso de matrizes de voxels, mas o que procedeu foi simplesmente manter o mesmo volume e aumentar o tamanho dos voxels dentro dele, logo, manter as mesmas dimensões gerais e diminuir a resolução, como pode ser visto nas imagens de exemplo ATUAL e PRETENDIDO (Fig. 7)
O mesmo acontece se o cálculo for aplicado nos demais eixos, teremos então os seguintes resultados para as dimensões do voxel: 0,01099 x 0,01099 x 0,01219 mm.
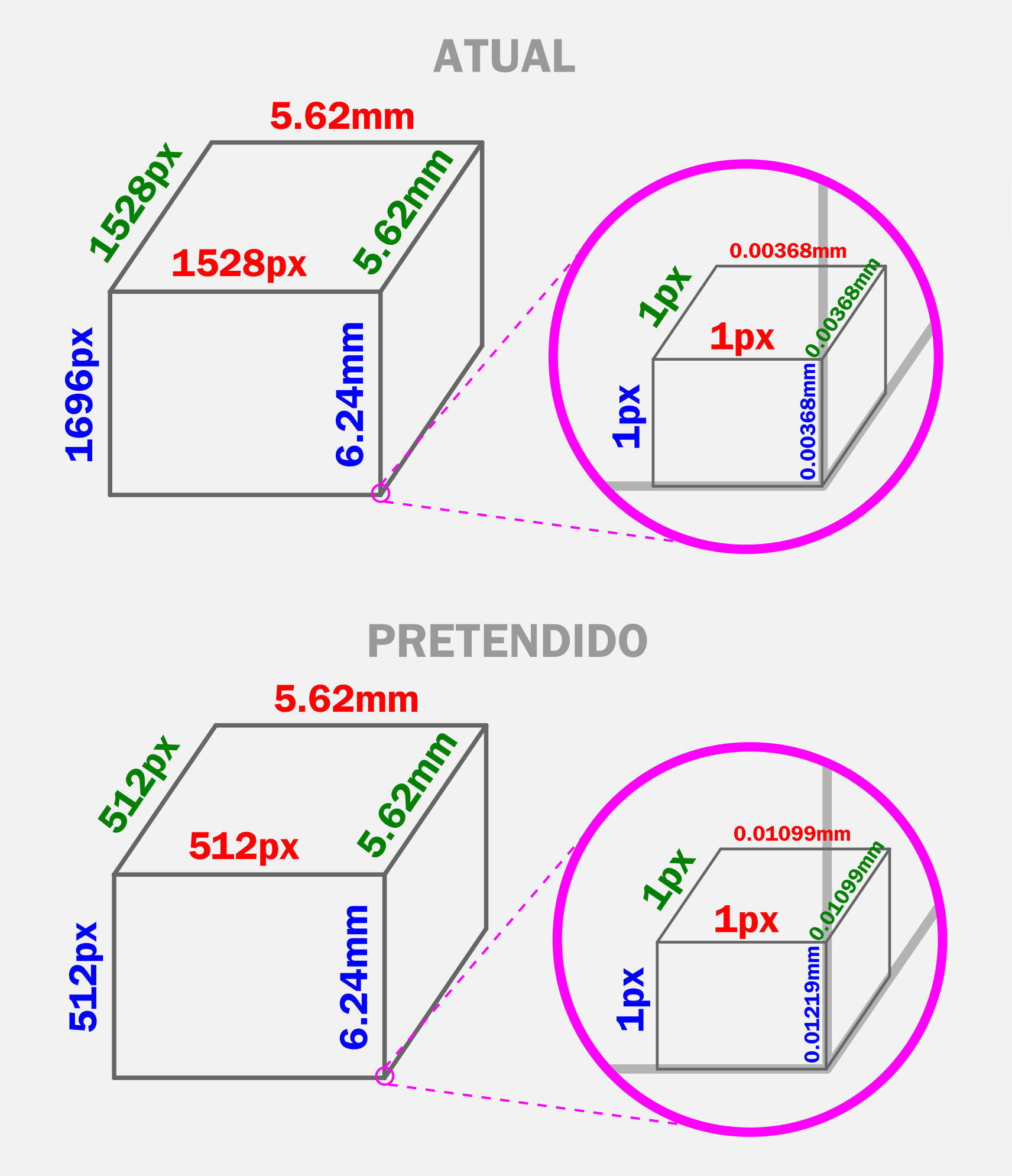
Dimensões em pixels e voxels.¶
Cabe reforçar que as dimensões da tomografia permanaceram as mesmas, ou seja: 5,62 mm x 5,62 mm x 6,24 mm. O que mudou foi o tamanho de cada voxel em milímetro. Anteriormente cada voxel contava com as seguintes dimensões: 0,00368 mm x 0,00368 mm x 0,00368. Depois do redimensionamento passaram a ter as seguintes dimensões: 0,01099 x 0,01099 x 0,01219 mm. Os pixels permaneceram os mesmos, ou seja 1 x 1 x 1 px (Fig. 8).
A diferença do último eixo se deveu a uma redução maior no mesmo, posto que anteriormente a matriz do voxel era de 1528 x 1528 x 1696 px, ou seja o último eixo continha um valor diferente. Já a mariz posterior igualou os três eixos em 512 x 512 x 512 px.
Aviso
A dimensão da imagem em pixels não está atrelada necessariamente a dimensão em milímetros da mesma, isso é definido pelas milímetros do voxel. A microtomografia descrita aqui é um bom exemplo, posto que a resolução das fatias era grande, mas as dimensões das mesmas são reduzidíssimas quando comparadas com a matriz de uma tomografia médica.
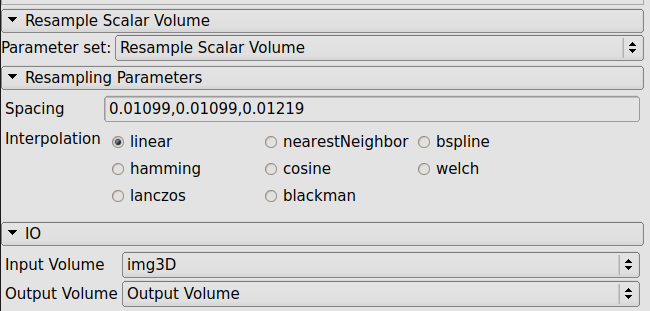
Redimensionamento.¶
Para mudar a resolução da tomografia no Slicer é necessário clicar no menu dos módulos, e em seguida Legacy –> Filtering –> Resample Scalar Volume. Em Spacing, preencher com as dimensões do voxel, usando o ponto como delimitador decimal e a vírgula para separar os valores, mantendo selecionada logo abaixo a opção linear (Fig. 9).
Em Input Volume é selecionada a tomografia que sofrerá a alteração e em Output Volume o usuário pode selecionar a opção de igual nome.
A clicar em Apply uma nova matriz com menor resolução será criada.
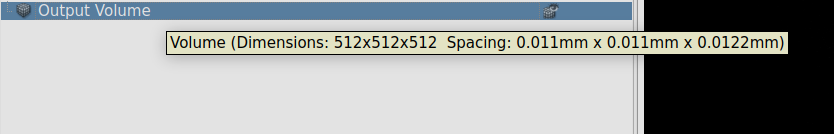
Redimensionamento.¶
Ao se observar as informações da tomografia gerada, atesta-se que a mesma conta agora com novas dimensões no voxel e foi reduzida para 512x512x512 pixels (Fig. 10).
Conclusão¶
A ferramenta de conversão de imagens em arquivos DICOM se mostrou rosbusta e funcional, principalmente quando utilizada em conjunto com a reconstrução de tomografias pela opção VTK ITK no OrtogOnBlender.
Galeria¶
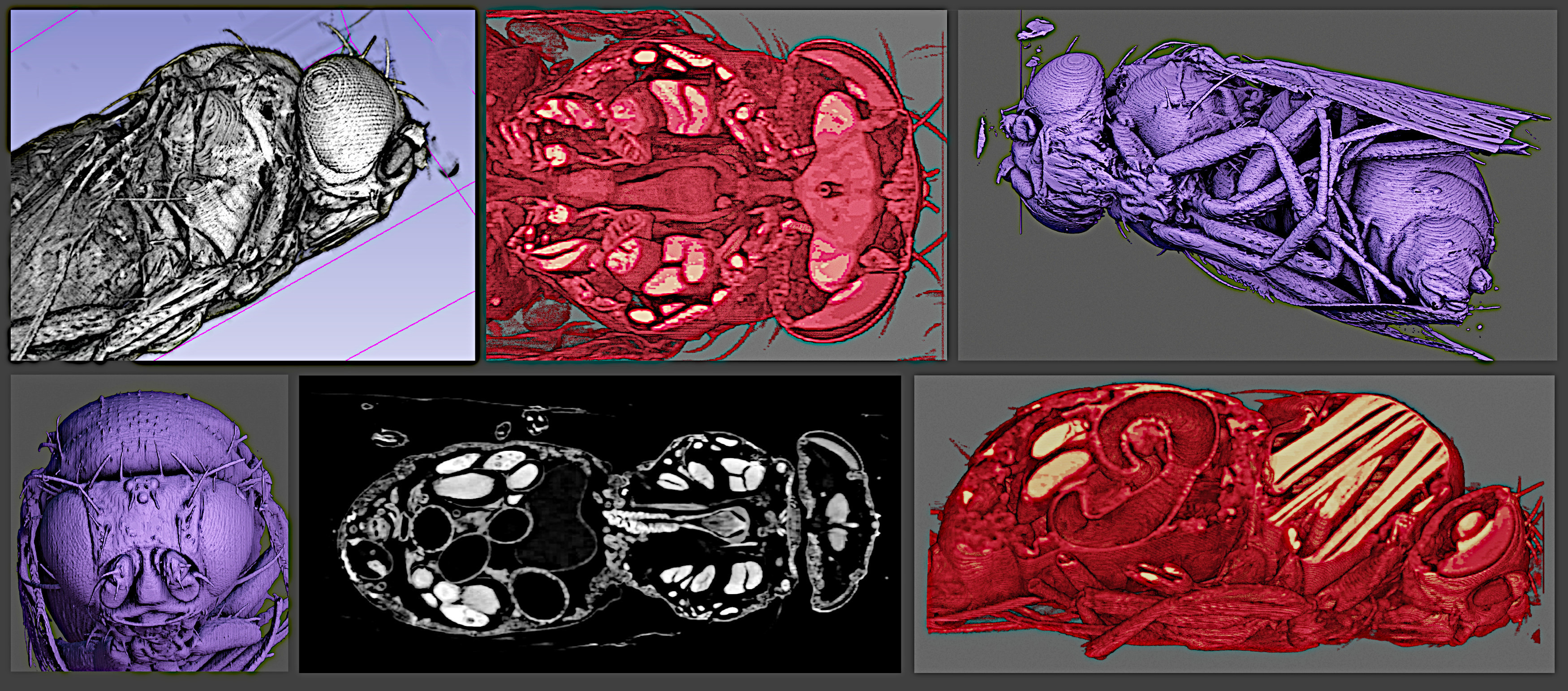
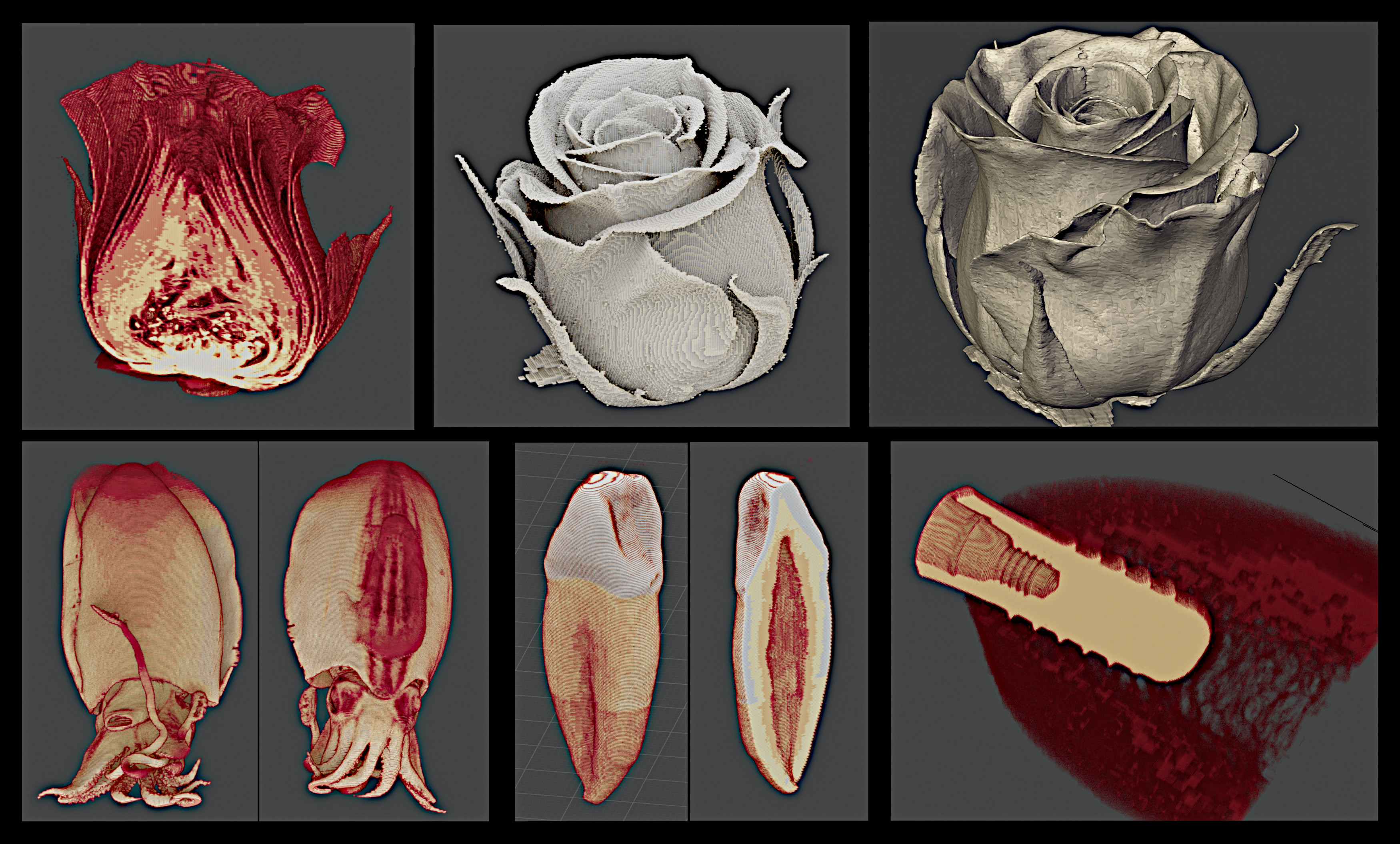
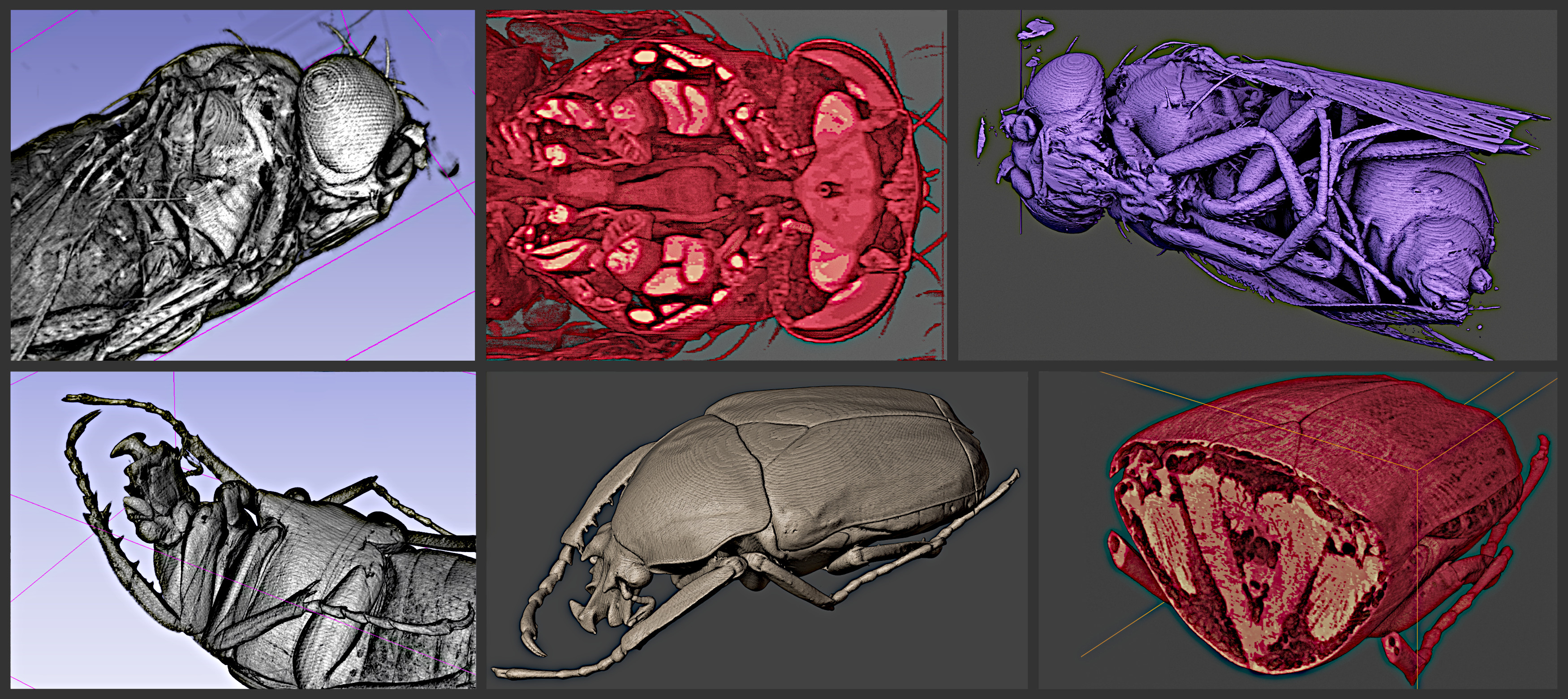
Alguns exemplos foram selecionados de modo a apresentar as possibilidades ofereceidas pelas ferramentas.
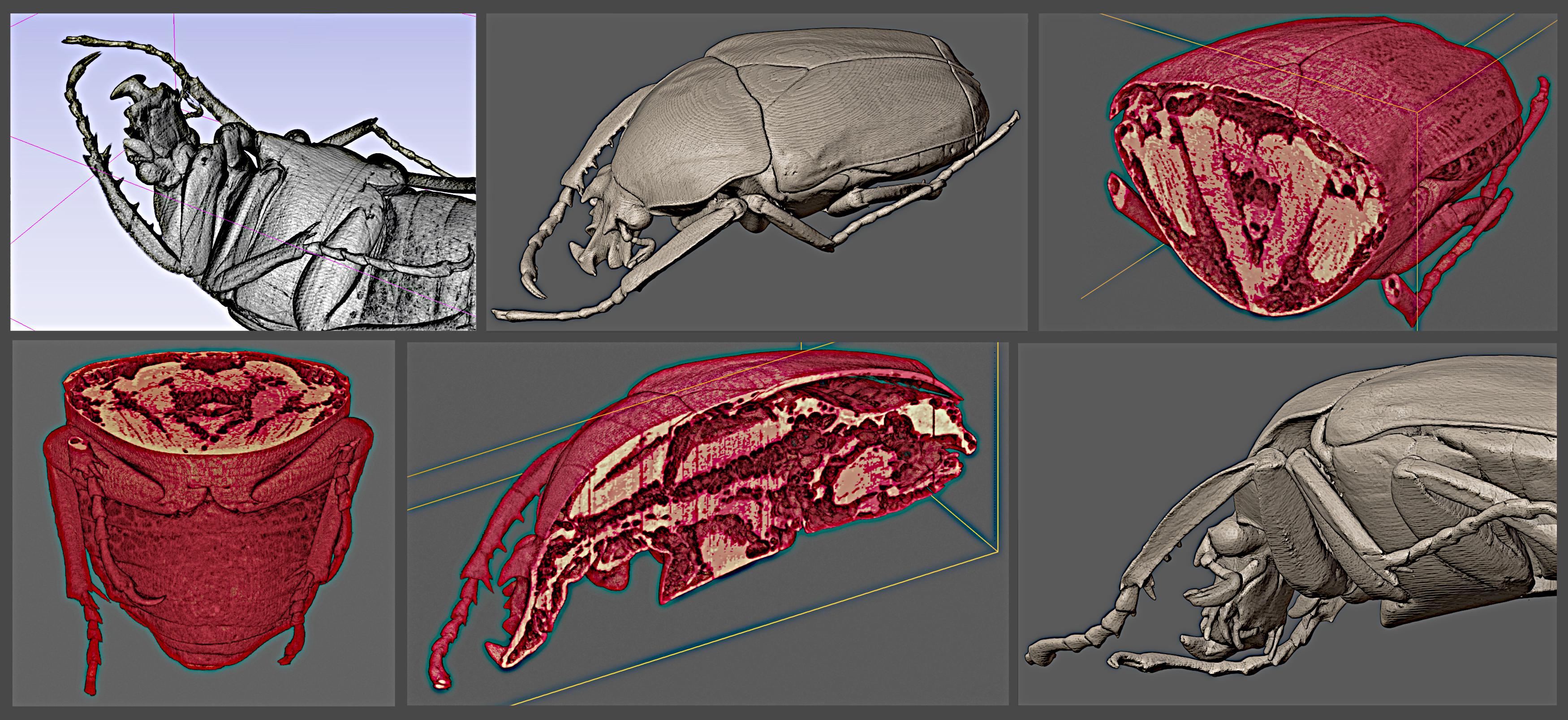
Microtomografia de um Dicronorrhina derbyana a partir de vídeo disponível na página da Universidade do Estado do Arizona.¶